Melanoma <- readRDS("../Poryecto_Genetica/Bases/Melanoma2.rds")Ejercicios clase EDA (genética)
Análisis exploratorio de datos
Introducción
Para más detalles revisar el capítulo 10 del compendio
El análisis exploratorio de datos (EDA) es una etapa fundamental en cualquier análisis de datos. Debe de ir antes de realizar cualquier prueba de hipótesis u otro análisis estadístico. El objetivo de esta etapa es explorar los datos para entender su estructura y distribución, identificar posibles problemas y errores, y detectar patrones y relaciones entre las variables. Además podemos formular hipótesis de cual es la realación entre las variables y probarlas en análisis posteriores.
EDA base Melanoma
Importar base de datos
- Cargue los datos del archivo
melanoma.rdsy muestre las primeras filas del dataframe con la funciónhead().
head(Melanoma) time status sex age year thickness ulcer
1 10 dead from other causes male 76 1972 6.76 presence
2 30 dead from other causes male 56 1968 0.65 absence
3 35 alive male 41 1977 1.34 absence
4 99 dead from other causes female 71 1968 2.90 absence
5 185 died from melanoma male 52 1965 12.08 presence
6 204 died from melanoma male 28 1971 4.84 presenceEstructura de las base de datos
- Visualización general de la variables y de los datos faltantes (si es que los hay)
str(Melanoma)'data.frame': 205 obs. of 7 variables:
$ time : int 10 30 35 99 185 204 210 232 232 279 ...
$ status : Factor w/ 3 levels "died from melanoma",..: 3 3 2 3 1 1 1 3 1 1 ...
$ sex : Factor w/ 2 levels "female","male": 2 2 2 1 2 2 2 1 2 1 ...
$ age : int 76 56 41 71 52 28 77 60 49 68 ...
$ year : int 1972 1968 1977 1968 1965 1971 1972 1974 1968 1971 ...
$ thickness: num 6.76 0.65 1.34 2.9 12.08 ...
$ ulcer : Factor w/ 2 levels "absence","presence": 2 1 1 1 2 2 2 2 2 2 ...# Si lo require instale:
install.packages("visdat")visdat::vis_dat(Melanoma)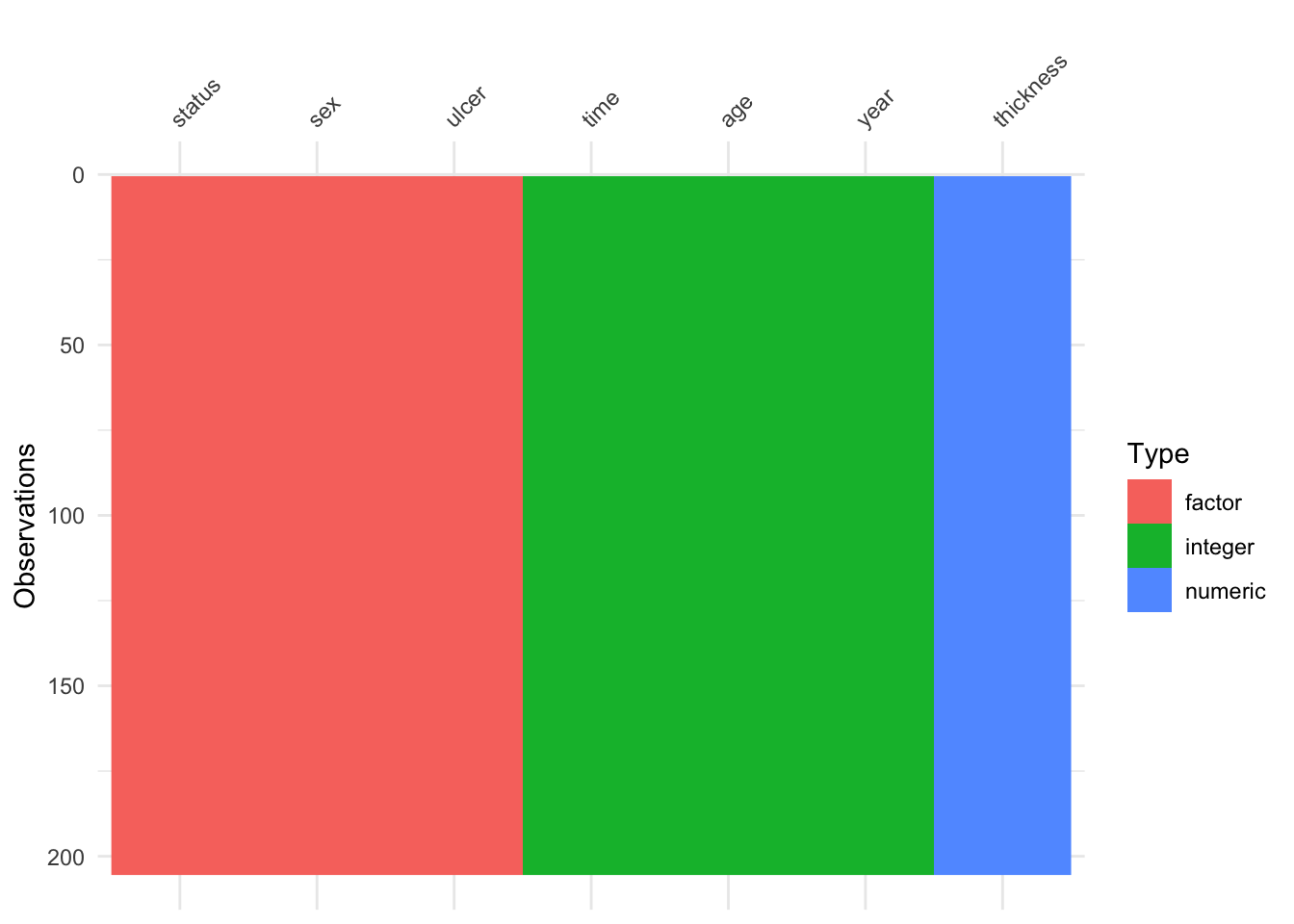
También podemos hacer la visualización de NA y aplicar un facet
visdat::vis_miss(Melanoma) # Para ver el % de valores perdidos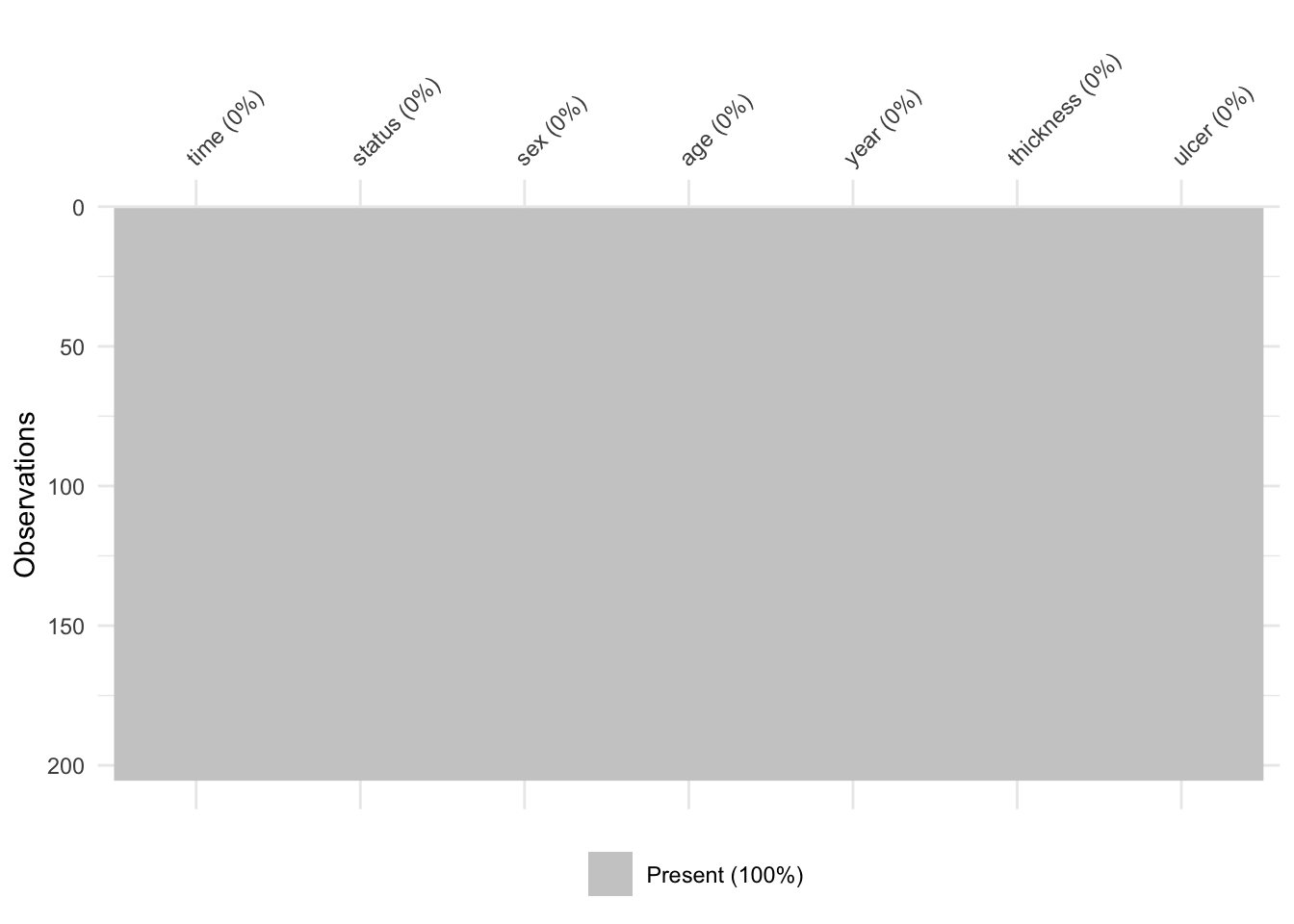
visdat::vis_dat(Melanoma, facet = status)
Errores, NA y datos atípicos
- Identificación de errores y datos atípicos
summary(Melanoma) time status sex age
Min. : 10 died from melanoma : 57 female:126 Min. : 4.00
1st Qu.:1525 alive :134 male : 79 1st Qu.:42.00
Median :2005 dead from other causes: 14 Median :54.00
Mean :2153 Mean :52.46
3rd Qu.:3042 3rd Qu.:65.00
Max. :5565 Max. :95.00
year thickness ulcer
Min. :1962 Min. : 0.10 absence :115
1st Qu.:1968 1st Qu.: 0.97 presence: 90
Median :1970 Median : 1.94
Mean :1970 Mean : 2.92
3rd Qu.:1972 3rd Qu.: 3.56
Max. :1977 Max. :17.42 library(tidyverse)── Attaching core tidyverse packages ──────────────────────── tidyverse 2.0.0 ──
✔ dplyr 1.1.4 ✔ readr 2.1.5
✔ forcats 1.0.0 ✔ stringr 1.5.1
✔ ggplot2 3.5.1 ✔ tibble 3.2.1
✔ lubridate 1.9.3 ✔ tidyr 1.3.1
✔ purrr 1.0.2
── Conflicts ────────────────────────────────────────── tidyverse_conflicts() ──
✖ dplyr::filter() masks stats::filter()
✖ dplyr::lag() masks stats::lag()
ℹ Use the conflicted package (<http://conflicted.r-lib.org/>) to force all conflicts to become errorsMelanoma|>
ggplot(aes(y = age))+
geom_boxplot()
Repetir la gráfica para el resto de las variables numéricas
Normalidad de las variables numéricas
- Evaluación de la normalidad de las variables numéricas
Melanoma|>
ggplot(aes(x = age))+
geom_density()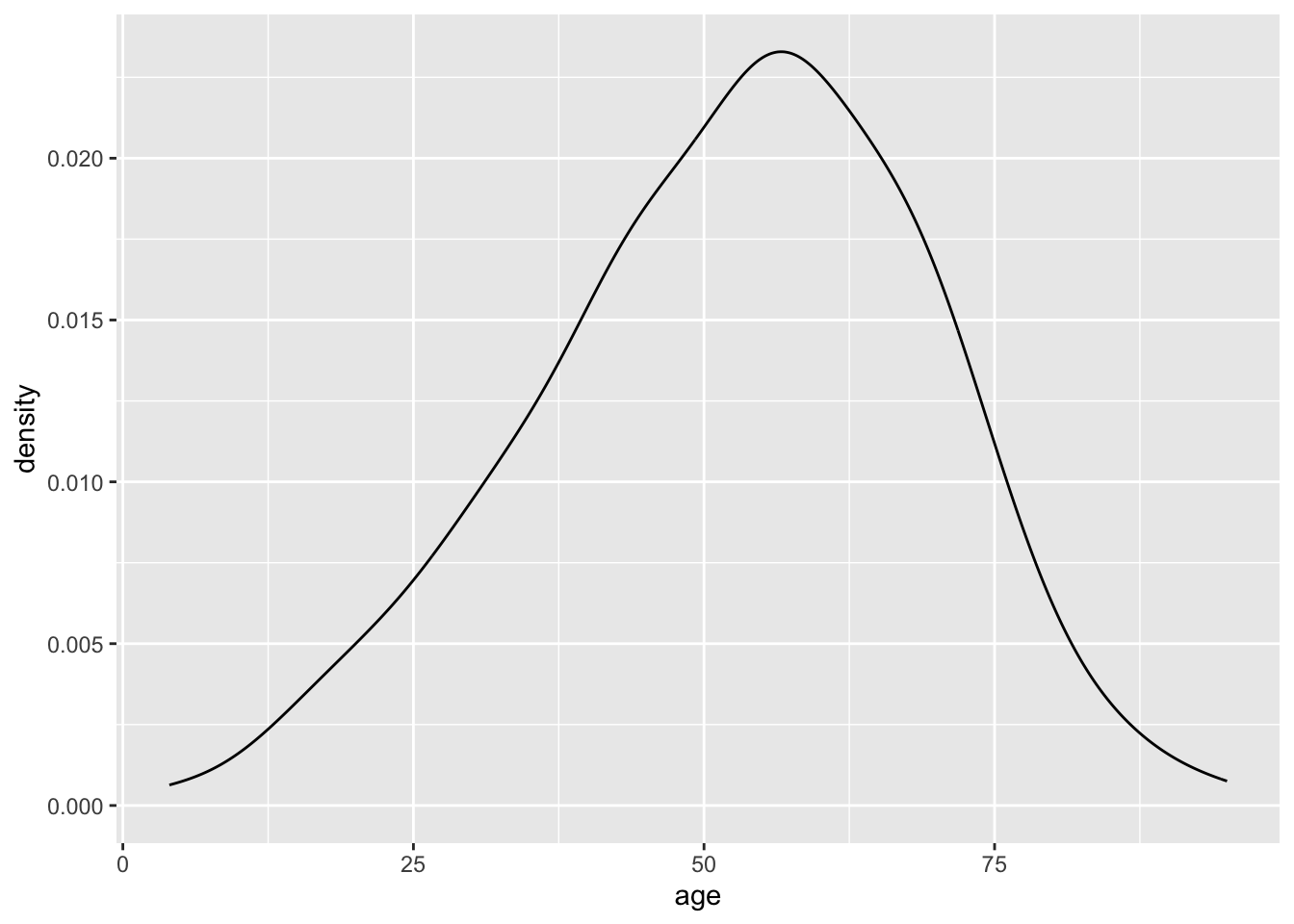
Repetir la gráfica para el resto de las variables numéricas
library(ggrain)Registered S3 methods overwritten by 'ggpp':
method from
heightDetails.titleGrob ggplot2
widthDetails.titleGrob ggplot2Melanoma|>
ggplot(aes(x = 1, y=age))+
geom_rain(fill = "cyan4", col="cyan4", alpha = 0.4)+
theme_linedraw()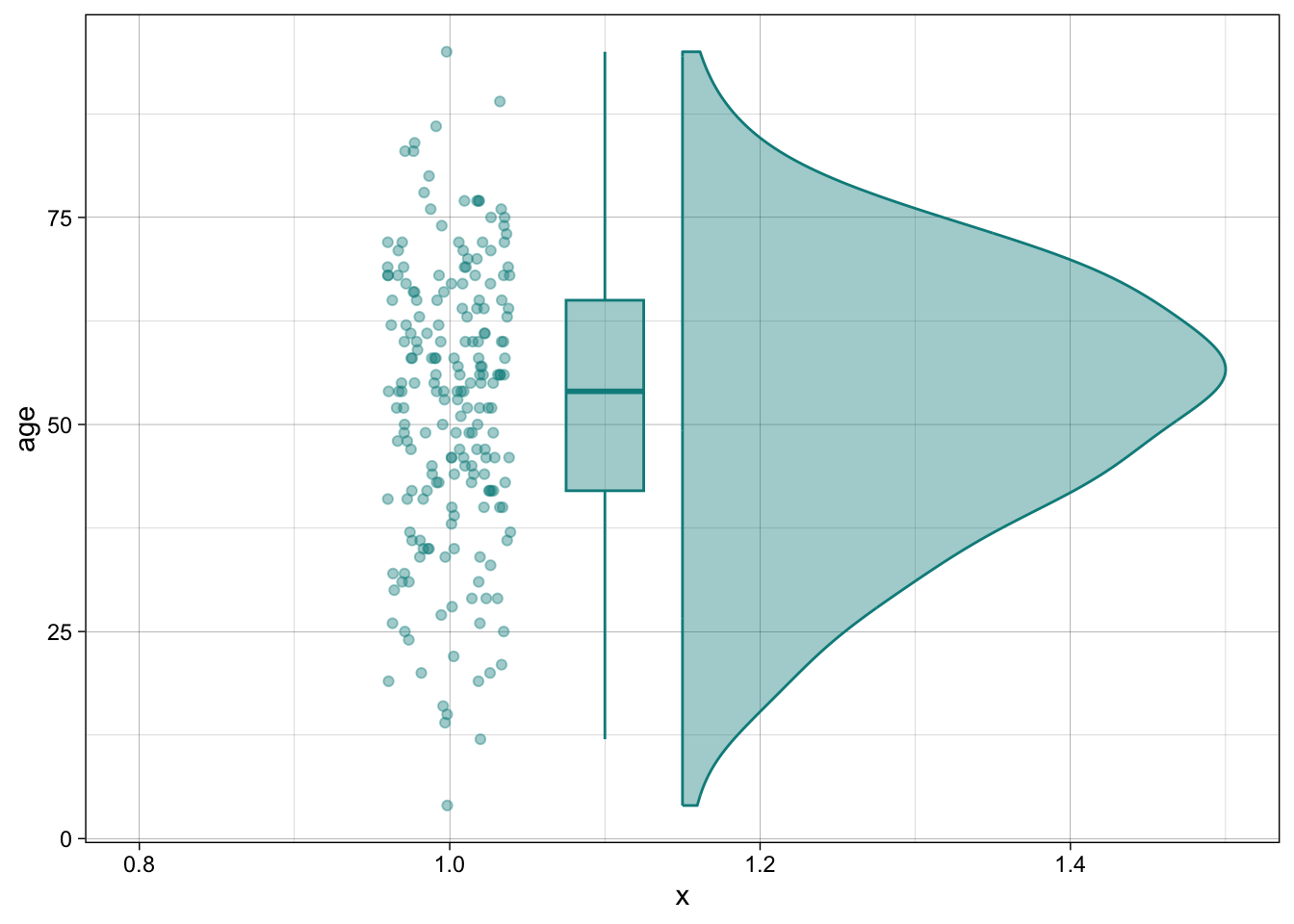
Repetir la gráfica para el resto de las variables numéricas
nortest::lillie.test(Melanoma$age)
Lilliefors (Kolmogorov-Smirnov) normality test
data: Melanoma$age
D = 0.068425, p-value = 0.0208Repetir para el resto de las variables numéricas
Exploración de las variables categóricas
- Exploración gráfica de las variables categóricas
Melanoma|>
ggplot(aes(x=status, fill=status))+
geom_bar()+
theme_linedraw()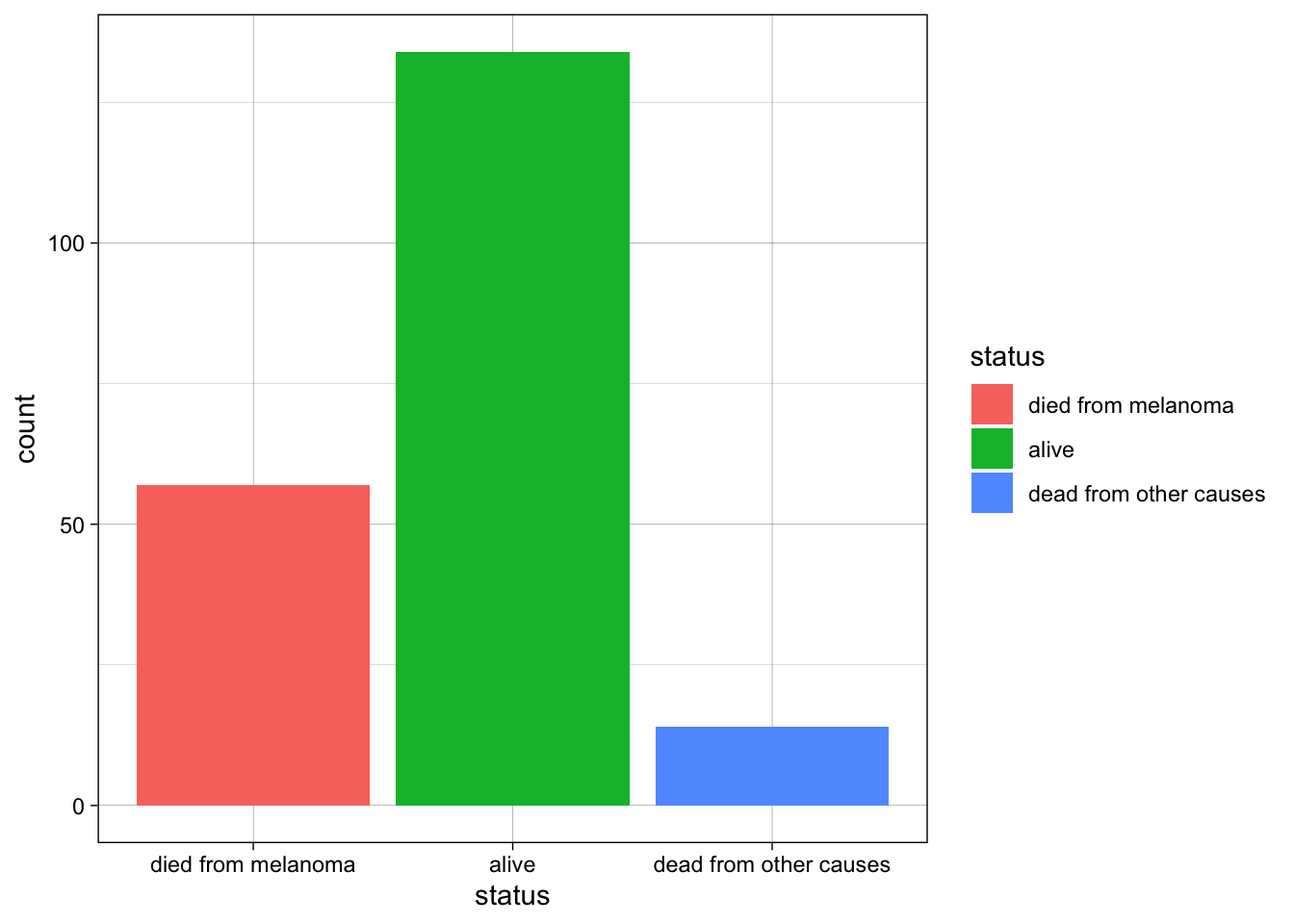
table(Melanoma$status)|>
prop.table()|>
barplot(ylab="Proporción", xlab="Status", col=c("#FCBA04", "#A50104",
"#590004"))
Repetir para el resto de las variables categóricas
Estadística descriptiva
summary(Melanoma) time status sex age
Min. : 10 died from melanoma : 57 female:126 Min. : 4.00
1st Qu.:1525 alive :134 male : 79 1st Qu.:42.00
Median :2005 dead from other causes: 14 Median :54.00
Mean :2153 Mean :52.46
3rd Qu.:3042 3rd Qu.:65.00
Max. :5565 Max. :95.00
year thickness ulcer
Min. :1962 Min. : 0.10 absence :115
1st Qu.:1968 1st Qu.: 0.97 presence: 90
Median :1970 Median : 1.94
Mean :1970 Mean : 2.92
3rd Qu.:1972 3rd Qu.: 3.56
Max. :1977 Max. :17.42 Para las variables cuantitativas podemos también usar las siguientes funciones:
- mean() # Para la media
- median() # Para la mediana
- sd() # Para la desviación estándar
- var() # Para la varianza
- range() # Para el rango
- quantile() # Para los cuantiles
- IQR() # Para el rango intercuartílico
En el caso de las variables categóricas podemos usar: - table() # Para la frecuencia - prop.table() # Para la proporción
Relación entre variables
- Relación entre variables
Nuestro objetivo que variables se relacionan con el status de los pacientes. Primero vamos a crear una nueva variable en la que vamos agrupar a los pacientes que murieron por otras causas y los que continúan vivos.
library(forcats)
Melanoma$status2 <- fct_collapse(
Melanoma$status,"No muerte por melanoma"=
c("alive","dead from other causes"))Una explicación más detallada del código anterior se explica a continuación:
# Carga la librería forcats, que proporciona herramientas para trabajar con factores
library(forcats)
# Utiliza la función fct_collapse() para combinar niveles de un factor en un nuevo nivel.
# En este caso, estamos creando una nueva variable llamada 'status2' en el dataframe 'Melanoma'.
Melanoma$status2 <- fct_collapse(
Melanoma$status, # Especifica el factor original, 'status', del dataframe 'Melanoma'.
# Define el nuevo nivel "No muerte por melanoma", que agrupa los niveles
# "alive" (vivo) y "dead from other causes" (muerto por otras causas).
"No muerte por melanoma" = c("alive", "dead from other causes")
)
# Como resultado, 'Melanoma$status2' tendrá un nuevo nivel "No muerte por melanoma"
# que combina los casos donde el paciente está vivo o murió por otras causas.Ahora vamos a explorar la relación entre las variables y el status de los pacientes, primero entre los tres grupos y después entre los dos grupos
Edad
Melanoma|>
ggplot(aes(x=status, y=age, fill=status))+
geom_boxplot()+
theme_classic()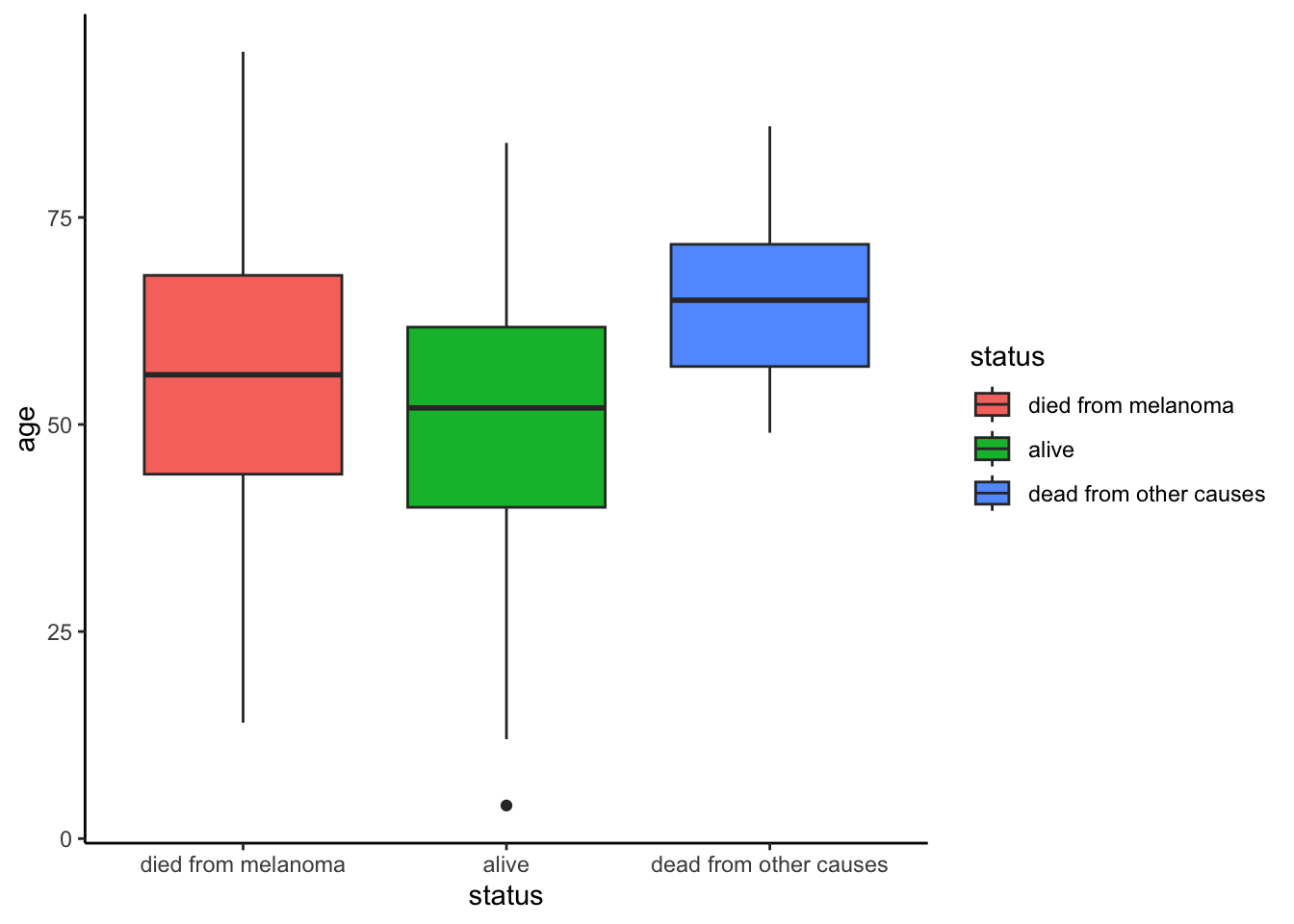
Melanoma|>
ggplot(aes(x=age, fill=status))+
geom_density(alpha=0.5)+
theme_classic()
Melanoma|>
ggplot(aes(x=1, y =age , fill=status))+
geom_rain(alpha=0.5)+
facet_grid(~status)+
theme_linedraw()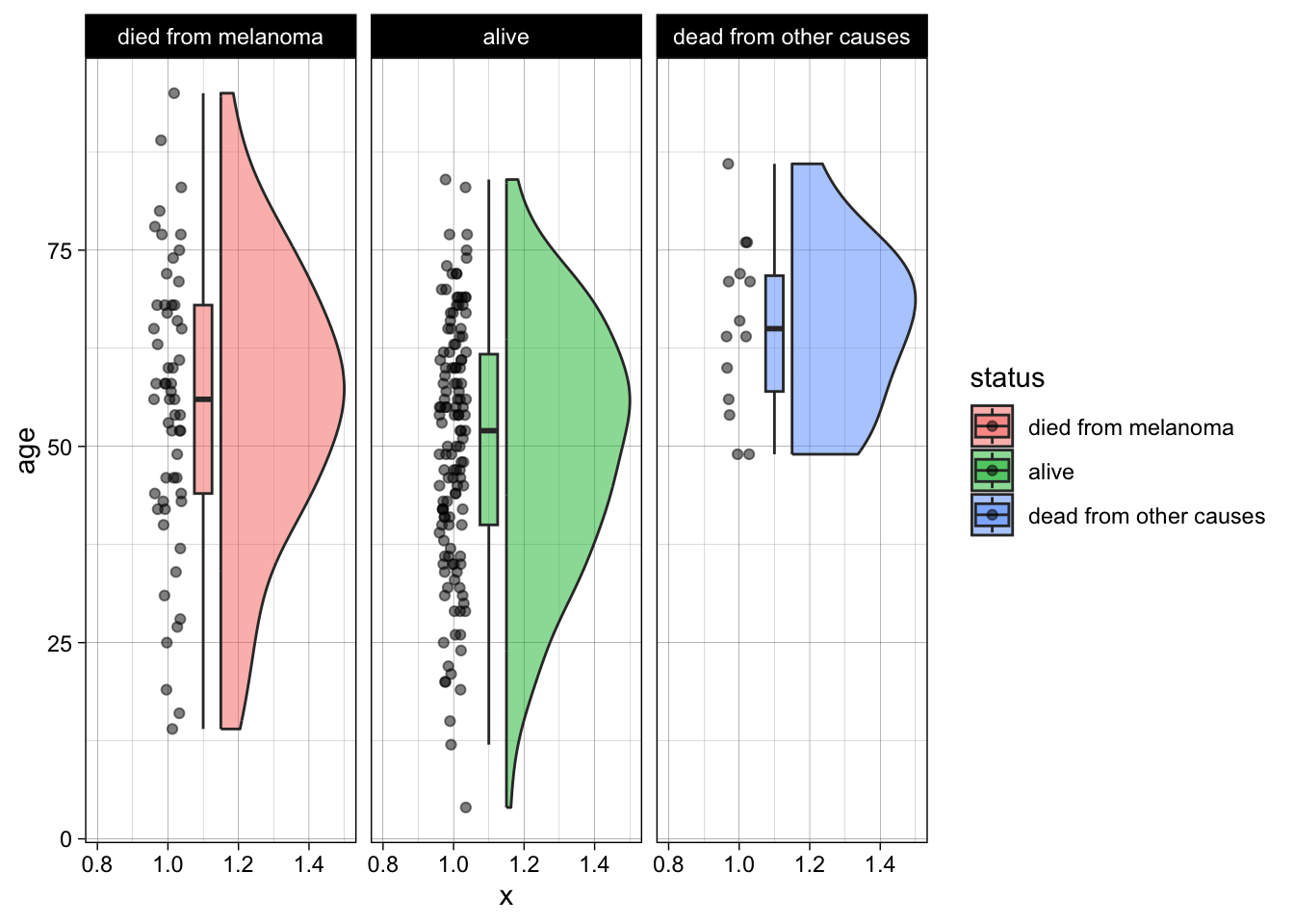
Tamaño del tumor
Melanoma|>
ggplot(aes(x=status, y=thickness, fill=status))+
geom_boxplot()+
theme_classic()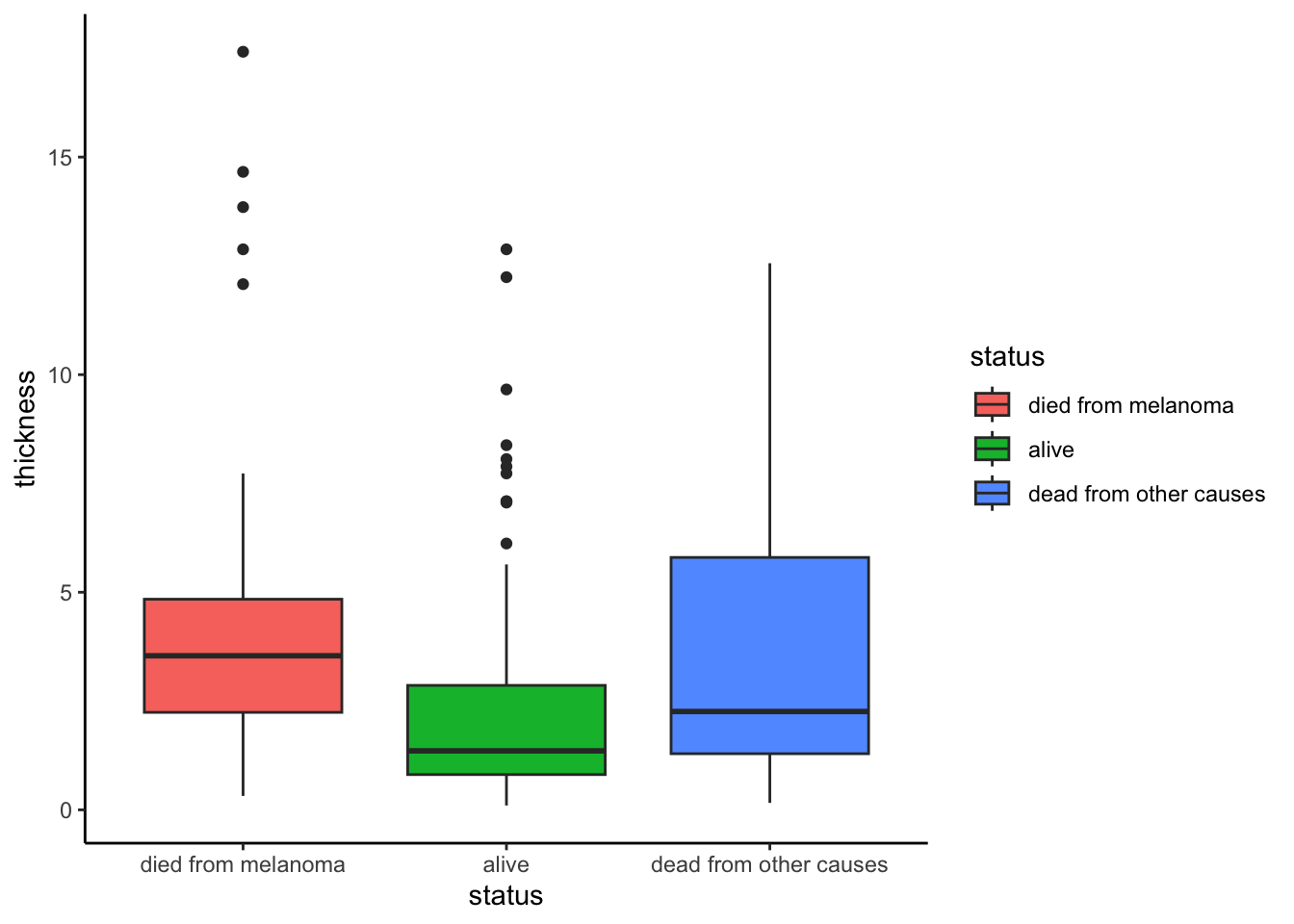
Melanoma|>
ggplot(aes(x=thickness, fill=status))+
geom_density(alpha=0.5)+
theme_classic()
Melanoma|>
ggplot(aes(x=1, y =thickness , fill=status))+
geom_rain(alpha=0.5)+
facet_grid(~status)+
theme_linedraw()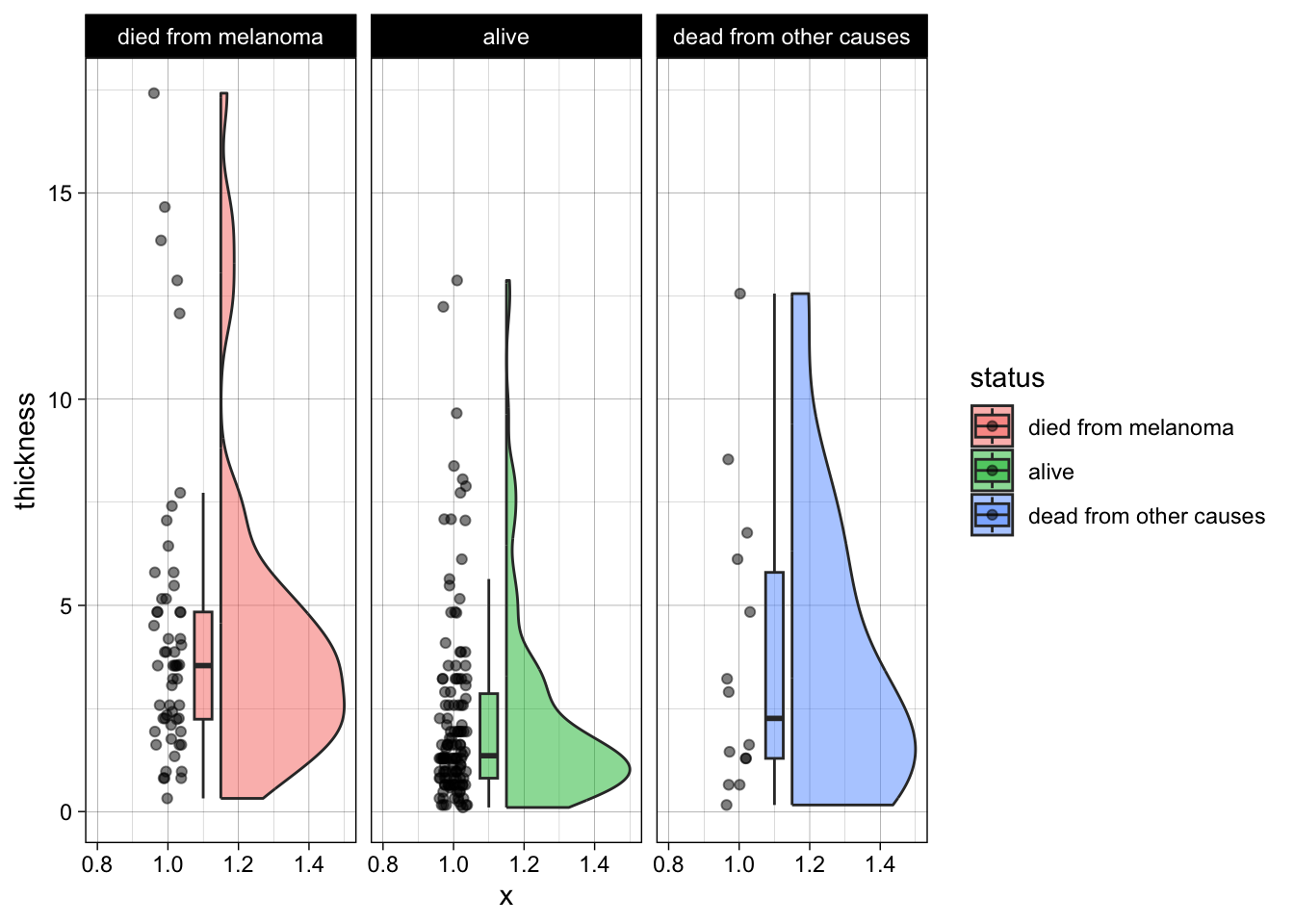
Sexo
Melanoma|>
ggplot(aes(x=status, fill=sex))+
geom_bar(position = "dodge")+
theme_bw()
Ulceras
Melanoma|>
ggplot(aes(x=status, fill=ulcer))+
geom_bar(position = "dodge")+
theme_bw()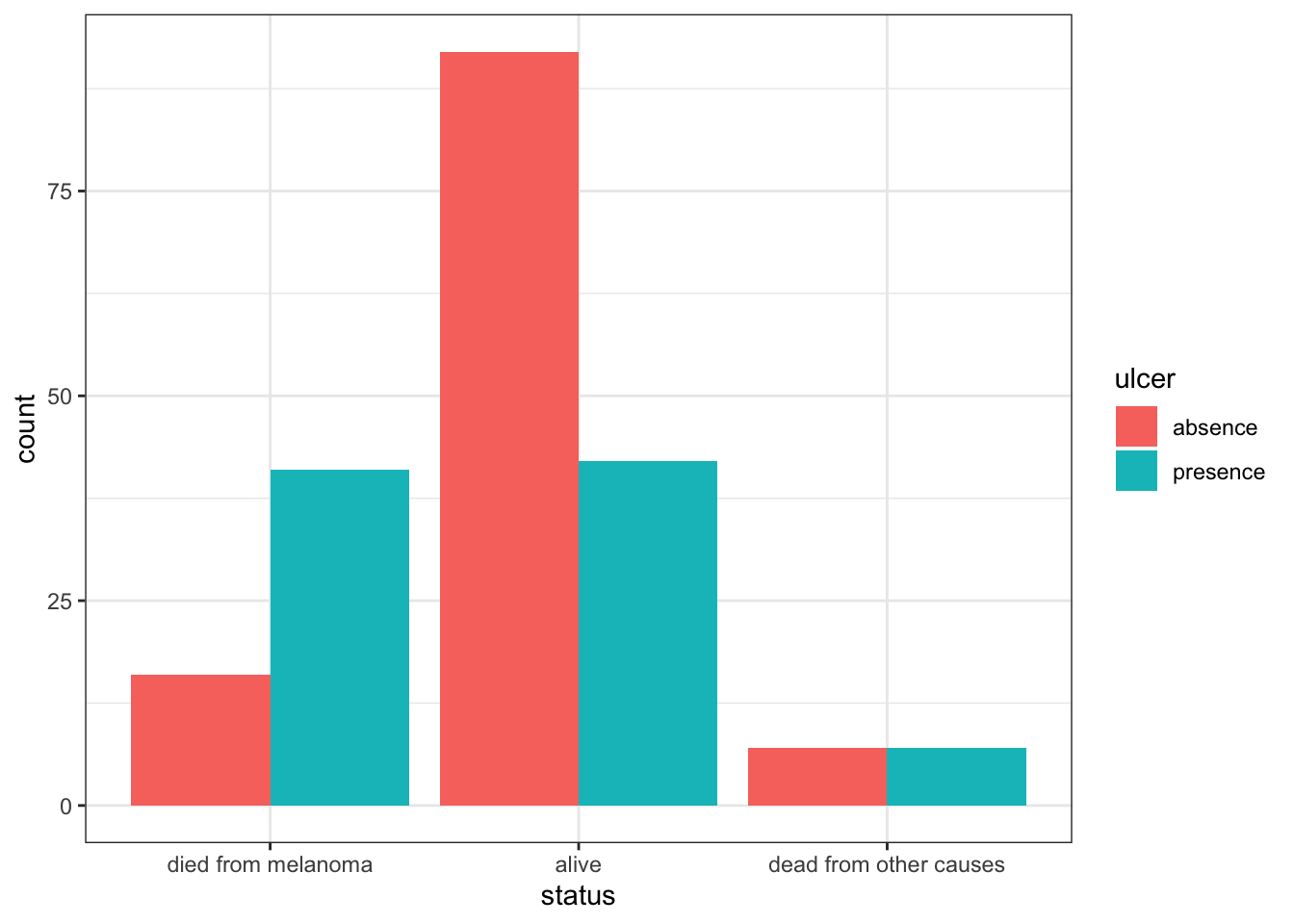
Repetir las gráficas para la variable status2
concluya y formule hipótesis anótelas
EDA con otras librerías
DataExplorer
install.packages("DataExplorer") ## En caso de ser necesario
DataExplorer::create_report(Melanoma)Ejercicios
Resuelva los ejercicios
- 10.7
- 10.8
- 10.9