Chapter 2 Modulos
Las bases de datos que fueron utilizadas para realizar el analisis de las variables de interes fueron las siguientes:
Modulo 64: Caracteristicas del hogar
RECH0: Informacion del hogar
RECH1: Cuestionario individual
RECH4: Cuestionario individual
Modulo 65: Caracteristicas de la vivienda
- RECH23: Informacion de la vivienda
Modulo 66: Indicadores basicos del MEF
- RECH0111: Informacion general
Modulo 70: Inmunizacion y salud
RECH43: Inmunizacion y salud
RECH85: Inmunizacion y salud
Modulo 74: Peso, Talla y Anemia
RECH5: Peso, Talla y Anemia en madres
RECH6: Peso, Talla y Anemia en niños
2.1 Bases de datos
library(tidyverse)
library(haven)
library(labelled)
library(skimr)
RECH0 <- read_sav("C:/Users/Bryan Fernandez/Desktop/Bryan/Lab Innovacion/ENDES/Modulo64/RECH0.SAV") %>%
mutate(HHID=str_trim(HHID))
RECH1 <- read_sav("C:/Users/Bryan Fernandez/Desktop/Bryan/Lab Innovacion/ENDES/Modulo64/RECH1.SAV") %>%
mutate(HHID=str_trim(HHID)) %>%
rename(ID=HVIDX)
RECH4 <- read_sav("C:/Users/Bryan Fernandez/Desktop/Bryan/Lab Innovacion/ENDES/Modulo64/RECH4.SAV") %>%
mutate(HHID=str_trim(HHID)) %>%
rename(ID = IDXH4)
RECH23 <- read_sav("C:/Users/Bryan Fernandez/Desktop/Bryan/Lab Innovacion/ENDES/Modulo65/RECH23.SAV") %>%
mutate(HHID = str_trim(HHID))
REC0111 <- read_sav("C:/Users/Bryan Fernandez/Desktop/Bryan/Lab Innovacion/ENDES/638-Modulo66/REC0111.SAV") %>%
distinct(HHID, .keep_all = T)
REC43 <- read_sav("C:/Users/Bryan Fernandez/Desktop/Bryan/Lab Innovacion/ENDES/638-Modulo70/REC43.SAV") %>%
mutate(CASEID = str_squish(CASEID)) %>%
separate(CASEID, c("HHID", "ID"), sep = " ", remove = F) %>%
mutate(ID = as.numeric(ID)) %>%
distinct(CASEID, .keep_all = T)
REC95 <- read_sav("C:/Users/Bryan Fernandez/Desktop/Bryan/Lab Innovacion/ENDES/638-Modulo70/REC95.SAV") %>%
mutate(CASEID = str_squish(CASEID)) %>%
separate(CASEID, c("HHID", "ID"), sep = " ", remove = F) %>%
mutate(ID = as.numeric(ID)) %>%
distinct(CASEID, .keep_all = T)
RECH5 <- read_sav("C:/Users/Bryan Fernandez/Desktop/Bryan/Lab Innovacion/ENDES/638-Modulo74/RECH5.SAV") %>%
mutate(HHID = str_squish(HHID)) %>%
rename(ID = HA0)
RECH6 <- read_sav("C:/Users/Bryan Fernandez/Desktop/Bryan/Lab Innovacion/ENDES/638-Modulo74/RECH6.SAV") %>%
mutate(HHID = str_squish(HHID)) %>%
rename(ID = HC0)
hh <- RECH0 %>%
full_join(RECH23, by = "HHID")
RH14 <- RECH1 %>%
full_join(RECH4, by = c("HHID", "ID"))2.1.1 Base final
El resultado final de todas las uniones se almacenara en el objeto mother
2.2 Exploracion de datos
Las variables de interes para el presente trabajo de investigacion son las siguientes:
HA57: Anemia
HA53: Nivel de hemoglobina en sangre (g-dl - 1 decimal)
HA2: Peso en kilogramos (1dec)
HA1: Edad en años
Ubigeo: codigo geografico
HV104: Sexo
HV024: Region (departamento)
HV025: Tipo de residencia (urbano y rural)
HV026: Tipo de ciudad (capital -> Campo)
HV109: Nivel educativo (Sin nivel -> Superior)
HV207: Su hogar ¿Tiene radio?
HV208: Su hogar ¿Tiene televisor?
HV225: Servicio higienico compartido
HV235: Ubicacion de la fuente de agua
HV241: Comida preparada en el hogar, edificio separado o fuera
HV242: Hay un cuarto para la cocina
SH42: Agua potable disponible todo el dia
SHREGION: Region natural (Lima metr -> Selva)
HA54: Actualmente embarazada
HV270: Indice de riqueza
S466GA: Practico como dar de lactar al niño/a
S466GB: Practico como preparar los alimentos al niño/a
S465DB_A: Ultimos 12 meses recibió del MINSA Hierro en jarabe
2.3 Prevalencia de anemia
library(tidyverse)
library(codebook)
library(table1)
library(viridis)
dat <- mother %>%
dplyr::select(HA57, HA1, ubigeo, HV104, HV024, HV025, HV026, HV109, HA54, SHREGION,
HV270,S466GA, S466GB,S470,S465DB_A, HV207, HV208, HV225, HV235, HV241, HV242, SH42, HA53, HA2) %>%
filter(!is.na(HA57)) %>%
mutate_at(c("HA57","HV104", "HV024", "HV025","HV026", "HV109", "HA54", "SHREGION",
"HV270","S466GA","S466GB","S470","S465DB_A","HV207","HV208", "HV225",
"HV235", "HV241", "HV242", "SH42"), .funs = haven::as_factor) %>%
mutate(anem = ifelse(HA57=="No anémico",0,1),
Anemic = factor(anem, levels = c(0,1) ,labels = c("No Anemic", "Anemic")))
table1(~ Anemic + HA57, data = dat)| Overall (n=19669) |
|
|---|---|
| Anemic | |
| No Anemic | 15501 (78.8%) |
| Anemic | 4168 (21.2%) |
| Nivel de Anemia | |
| Severa | 20 (0.1%) |
| Moderada | 453 (2.3%) |
| Leve | 3695 (18.8%) |
| No anémico | 15501 (78.8%) |
| No Anemic (n=15501) |
Anemic (n=4168) |
Overall (n=19669) |
|
|---|---|---|---|
| Edad de la mujer en años | |||
| Mean (SD) | 30.4 (7.01) | 30.3 (7.42) | 30.4 (7.10) |
| Median [Min, Max] | 30.0 [13.0, 49.0] | 30.0 [14.0, 49.0] | 30.0 [13.0, 49.0] |
| Sexo | |||
| Hombre | 0 (0%) | 0 (0%) | 0 (0%) |
| Mujer | 15501 (100%) | 4168 (100%) | 19669 (100%) |
| Región | |||
| Amazonas | 680 (4.4%) | 123 (3.0%) | 803 (4.1%) |
| Ancash | 568 (3.7%) | 111 (2.7%) | 679 (3.5%) |
| Apurimac | 551 (3.6%) | 112 (2.7%) | 663 (3.4%) |
| Arequipa | 551 (3.6%) | 157 (3.8%) | 708 (3.6%) |
| Ayacucho | 640 (4.1%) | 147 (3.5%) | 787 (4.0%) |
| Cajamarca | 625 (4.0%) | 81 (1.9%) | 706 (3.6%) |
| Callao | 546 (3.5%) | 173 (4.2%) | 719 (3.7%) |
| Cusco | 504 (3.3%) | 126 (3.0%) | 630 (3.2%) |
| Huancavelica | 480 (3.1%) | 177 (4.2%) | 657 (3.3%) |
| Huanuco | 667 (4.3%) | 120 (2.9%) | 787 (4.0%) |
| Ica | 571 (3.7%) | 168 (4.0%) | 739 (3.8%) |
| Junin | 569 (3.7%) | 173 (4.2%) | 742 (3.8%) |
| La Libertad | 573 (3.7%) | 130 (3.1%) | 703 (3.6%) |
| Lambayeque | 668 (4.3%) | 123 (3.0%) | 791 (4.0%) |
| Lima | 1911 (12.3%) | 574 (13.8%) | 2485 (12.6%) |
| Loreto | 524 (3.4%) | 258 (6.2%) | 782 (4.0%) |
| Madre de Dios | 509 (3.3%) | 137 (3.3%) | 646 (3.3%) |
| Moquegua | 493 (3.2%) | 155 (3.7%) | 648 (3.3%) |
| Pasco | 475 (3.1%) | 147 (3.5%) | 622 (3.2%) |
| Piura | 634 (4.1%) | 154 (3.7%) | 788 (4.0%) |
| Puno | 338 (2.2%) | 179 (4.3%) | 517 (2.6%) |
| San Martin | 576 (3.7%) | 163 (3.9%) | 739 (3.8%) |
| Tacna | 550 (3.5%) | 156 (3.7%) | 706 (3.6%) |
| Tumbes | 656 (4.2%) | 131 (3.1%) | 787 (4.0%) |
| Ucayali | 642 (4.1%) | 193 (4.6%) | 835 (4.2%) |
| Área de residencia | |||
| Urbana | 10866 (70.1%) | 2921 (70.1%) | 13787 (70.1%) |
| Rural | 4635 (29.9%) | 1247 (29.9%) | 5882 (29.9%) |
| Lugar de residencia | |||
| Capital, ciudad grande | 1908 (12.3%) | 575 (13.8%) | 2483 (12.6%) |
| Ciudad pequeña | 4650 (30.0%) | 1195 (28.7%) | 5845 (29.7%) |
| Pueblo | 4308 (27.8%) | 1151 (27.6%) | 5459 (27.8%) |
| Campo | 4635 (29.9%) | 1247 (29.9%) | 5882 (29.9%) |
2.4 Tendencias
Prevalencia de anemia en el Peru segun tipo de ciudad
dat %>%
group_by(ubigeo, HV026) %>%
summarise(an = mean(anem, na.rm = T)) %>%
ggplot(aes(x = an, fill = HV026)) +
geom_density(alpha = .3) +
scale_fill_viridis_d() +
geom_vline(data = dat %>% group_by(HV026, ubigeo) %>% summarise(m = mean(anem, na.rm = T)) %>% ungroup() %>% group_by(HV026) %>% summarise(m = mean(m, na.rm = T)),
aes(xintercept = m, col = HV026), size = 1, linetype = "dashed") +
scale_color_viridis_d() +
labs(fill = "Area", col = "Area", x = "Prevalencia de anemia", y = "Densidad")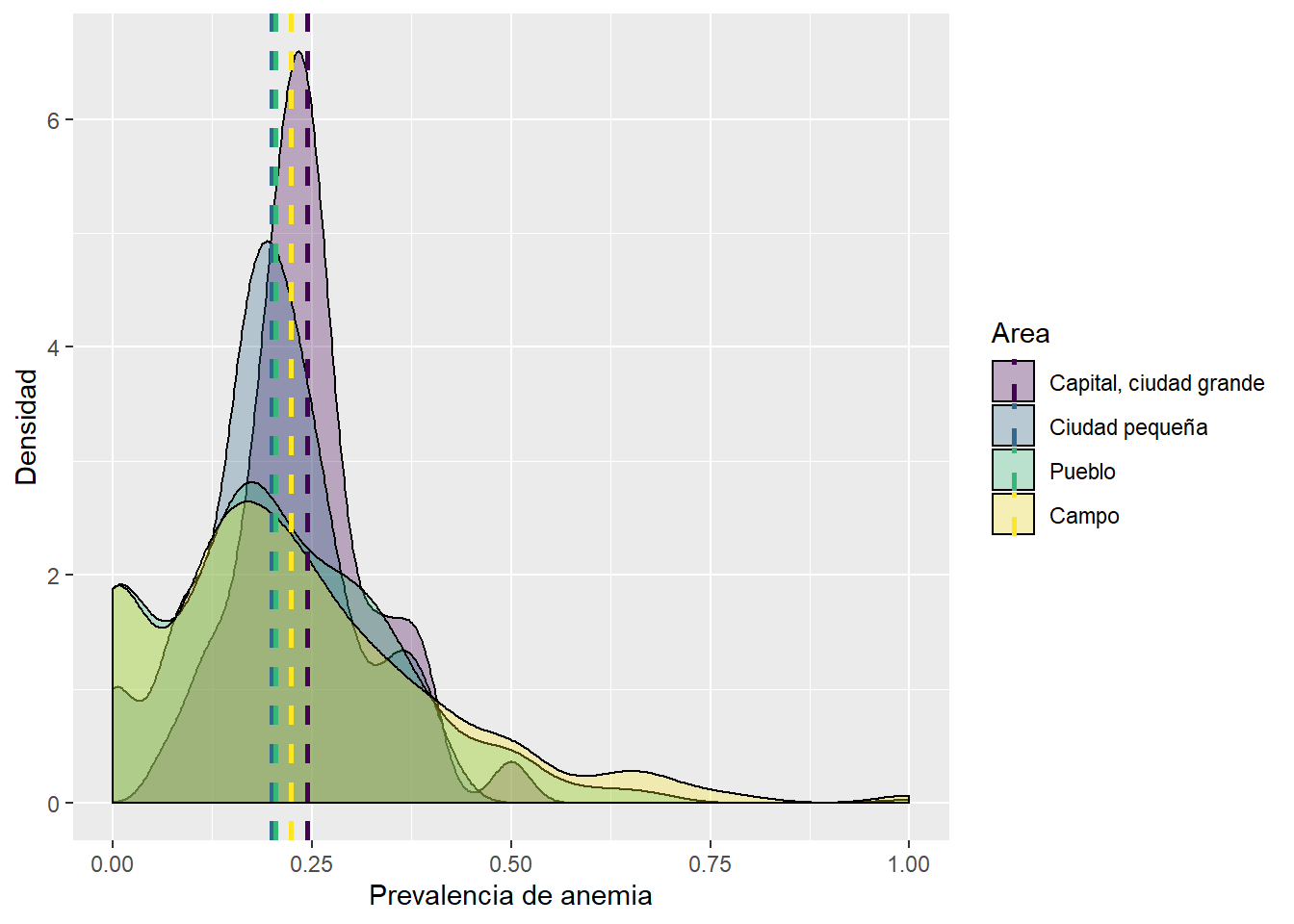
El grafico mostrado anteriormente, evidencia que las madres que se encuentran viviendo en la capital registran mayor prevalencia de anemia para el año 2018 en el Peru, mientras que la menor prevalencia de anemia se encuentra en las ciudades pequeñas.
Prevalencia de anemia segun tipo de residencia
dat %>%
group_by(ubigeo,HV025) %>%
summarise(an = mean(anem, na.rm = T)) %>%
ggplot(aes(x = an, fill = HV025)) +
geom_density(alpha = .3) +
scale_fill_viridis_d(option = "plasma") +
geom_vline(data = dat %>% group_by(HV025, ubigeo) %>% summarise(m = mean(anem, na.rm = T)) %>% ungroup() %>% group_by(HV025) %>% summarise(m = mean(m, na.rm = T)),
aes(xintercept = m, col = HV025), size = 1, linetype = "dashed", alpha = 0.3) +
scale_color_viridis_d(option = "plasma") +
labs(fill = "Area", col = "Area", x = "Prevalencia de anemia", y = "Densidad")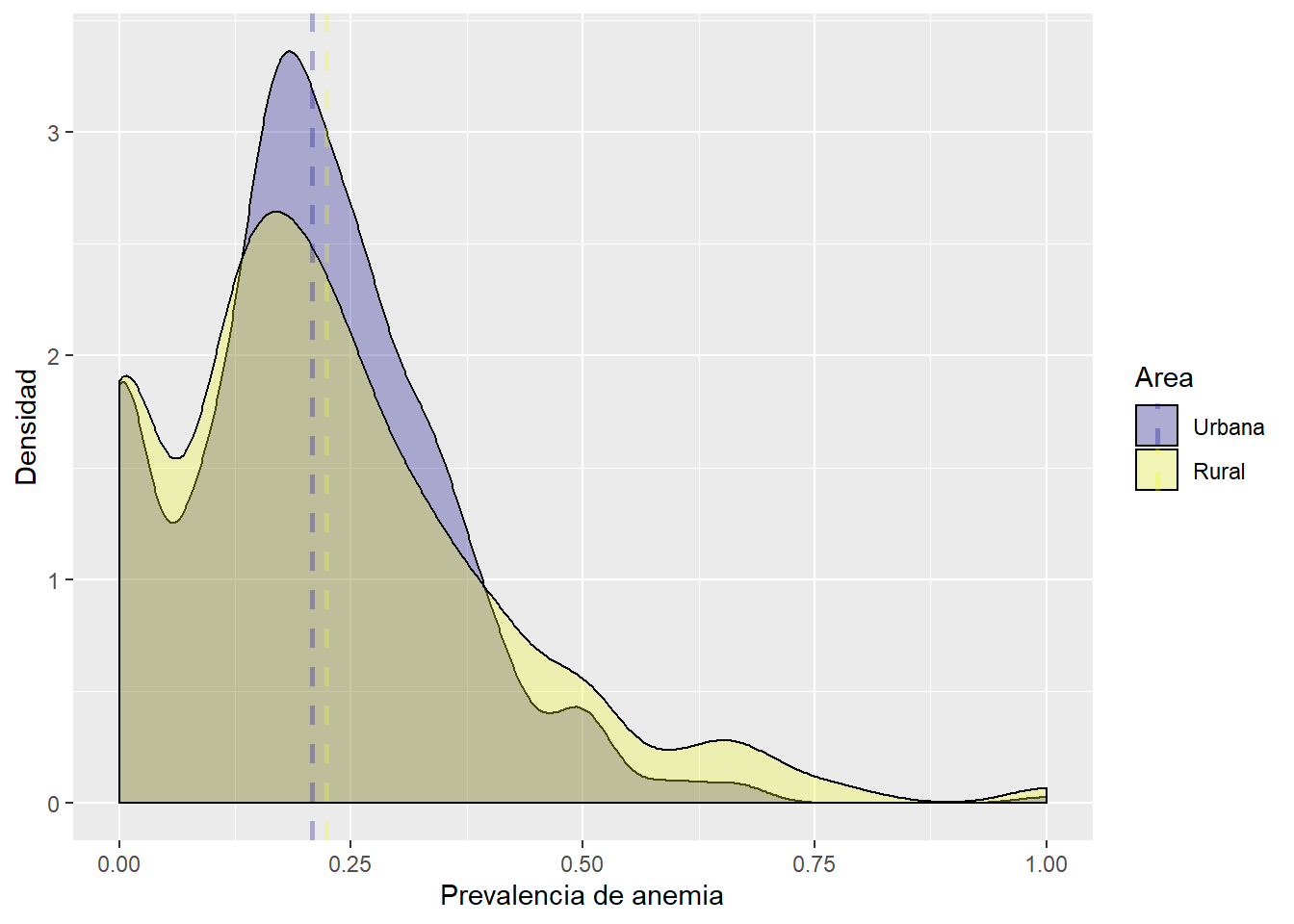
El grafico mostrado revela que las madres que se encuentran en el area rural presentan una mayor prevalencia de anemia que las personas que residen en el area urbana.
Prevalencia de anemia segun region
## PLOTLY grafico dinamico
dat %>%
group_by(ubigeo,HV024) %>%
summarise(an = mean(anem, na.rm = T)) %>%
ggplot(aes(x = an, fill = HV024)) +
geom_density(alpha = .2) +
scale_fill_viridis_d() +
#geom_vline(data = dat %>% group_by(HV024, ubigeo) %>% summarise(m = mean(anem, na.rm = T)) %>% ungroup() %>% group_by(HV024) %>% summarise(m = mean(m, na.rm = T)),
#aes(xintercept = m, col = HV024), size = 1, linetype = "dashed", alpha = 0.3) +
labs(fill = "Area", col = "Area", x = "Prevalencia de anemia", y = "Densidad") 
Prevalencia de anemia segun region natural
dat %>%
group_by(ubigeo,SHREGION) %>%
summarise(an = mean(anem, na.rm = T)) %>%
ggplot(aes(x = an, fill = SHREGION)) +
geom_density(alpha = .3) +
scale_fill_viridis_d() +
geom_vline(data = dat %>% group_by(SHREGION, ubigeo) %>% summarise(m = mean(anem, na.rm = T)) %>% ungroup() %>% group_by(SHREGION) %>% summarise(m = mean(m, na.rm = T)),
aes(xintercept = m, col = SHREGION), size = 1, linetype = "dashed", alpha = 0.5) +
scale_color_viridis_d() +
labs(fill = "Area", col = "Area", x = "Anemia prevalence") 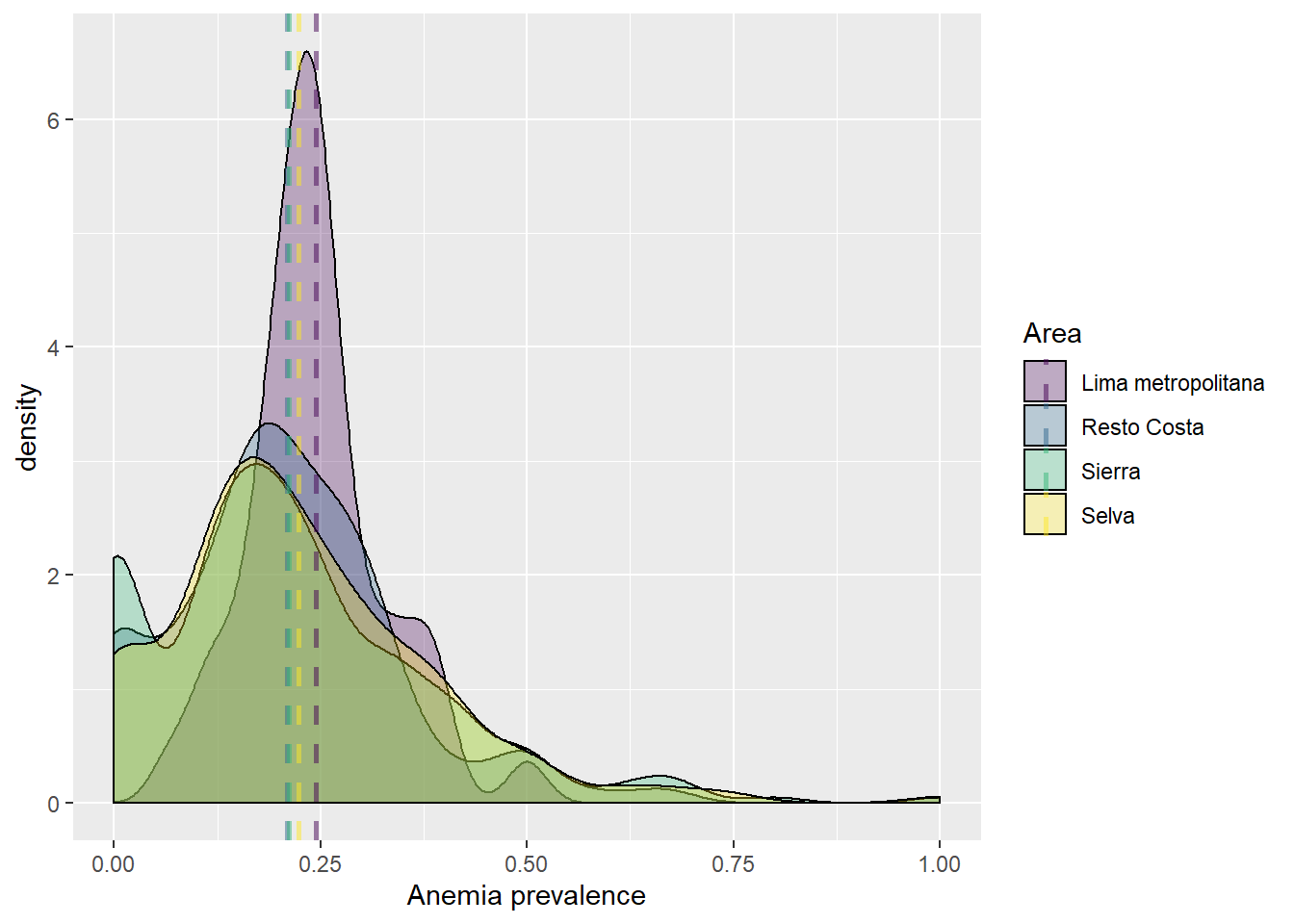
Para el año 2018, Lima Metropolitana se consolido como la region natural con mayor prevalencia de anemia en el Peru.
Prevalencia de anemia y edad segun sexo
dat %>%
group_by(HV104, HA1) %>%
summarise(an = mean(anem, na.rm = T)) %>%
ggplot(aes(y=an, x = HA1, fill = HV104, col = HV104, group = HV104)) +
geom_smooth() +
scale_fill_viridis_d() +
scale_color_viridis_d() +
labs(fill = "Sexo", col = "Sexo", y = "Prevalencia de anemia", x = "Edad en años") +
theme_bw()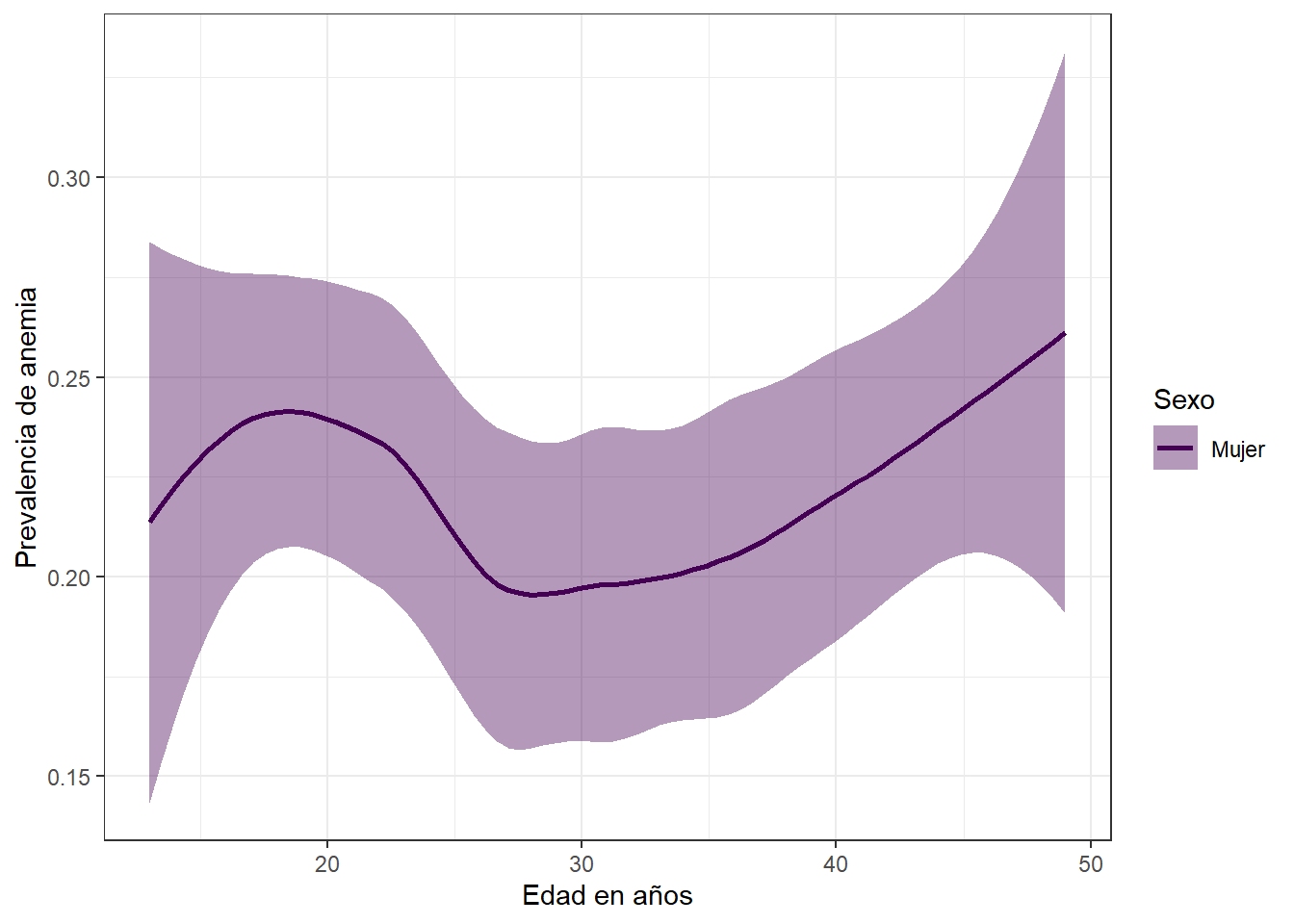
La prevalencia de anemia en madres tiene una tendencia marcada, incrementa a medida que pasa el tiempo, premisa que el grafico comprueba y ademas enos muestra que existe una disminucion de la prevalencia de dicha enfermedad entre los 20 y 25 años.
Prevalencia de anemia, edad y sexo segun tipo de residencia
dat %>%
group_by(HA1, HV025) %>%
summarise(an = mean(anem, na.rm = T)) %>%
ggplot(aes(y=an, x = HA1, fill = HV025, col = HV025, group = HV025)) +
geom_smooth() +
scale_fill_viridis_d() +
scale_color_viridis_d() +
labs(fill = "Area", col = "Area", y = "Prevalencia de anemia", x = "Edad en años") +
#facet_grid(.~HV025) +
theme_bw()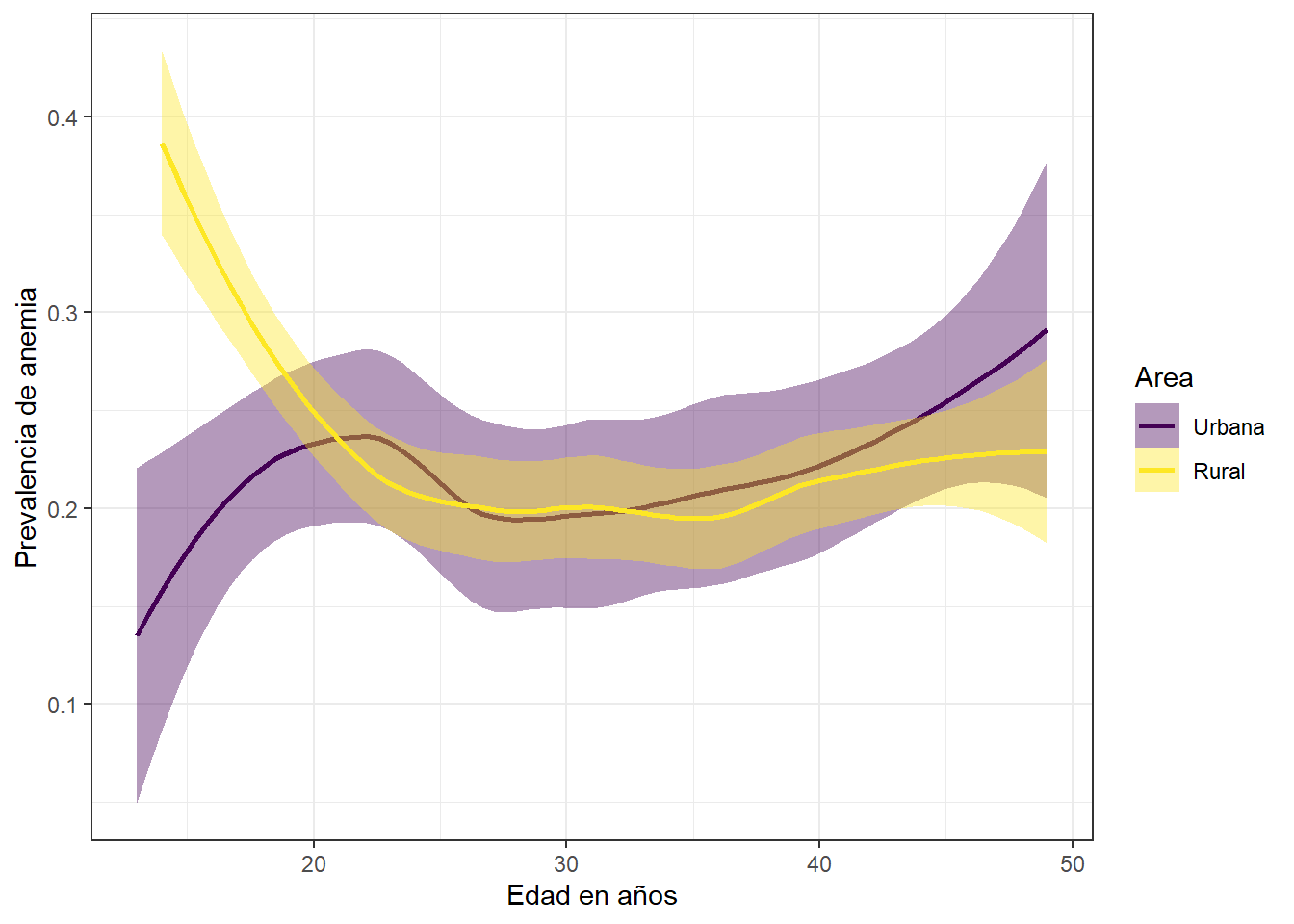
La prevalencia de anemia en madres, para el 2018, tuvo resultados opuestos para la primera etapa de vida de estas, ya que, para el area rural se evidencia una disminucion desde el año 0 hasta los 20 años aproximadamente. Mientras que para el area urbana, se logra apreciar un incremento de la prevalencia de anemia desde el año 0 hasta los 20 años. Para la etapa que esta comprendida entre los años 40 y 50, la prevalencia es mucho mayor en las madres que se encuentran en el area urbana.
Prevalencia de anemia, edad y sexo segun tipo de ciudad
dat %>%
group_by(HA1, HV025, HV026) %>%
summarise(an = mean(anem, na.rm = T)) %>%
ggplot(aes(y=an, x = HA1, fill = HV025, col = HV025, group = HV025)) +
geom_smooth() +
scale_fill_viridis_d() +
scale_color_viridis_d() +
labs(fill = "Area", col = "Area", y = "Prevalencia de anemia", x = "Edad en años") +
facet_grid(.~HV026) +
theme_bw()
Este grafico muestra la prevalencia de anemia de acuerdo al tipo de residencia y al tipo de area. La tendencia en el area urbana apunta al incremento de la prevalencia de anemia a medida que pasan los años (Capital, ciudad grande y pueblo), mientras que en el area rural, la tendencia apunta a su disminucion (campo).
colfunc<-colorRampPalette(c("#4bb9c3","#88d78c", "#d3d483", "#2b8282"))
plot(rep(1,100),col=(colfunc(100)), pch=19,cex=2)
dat %>%
group_by(ubigeo, HV026, HV025) %>%
summarise(an = mean(anem, na.rm = T)) %>%
ggplot(aes(x = HV026, y = an)) +
geom_violin(aes(fill = HV026)) +
geom_jitter(width = .2) +
geom_boxplot(fill = NA) +
labs(x = "Tipo de ciudad", y = "Prevalencia de anemia") +
geom_hline(aes(yintercept=median(an,na.rm = T)),
color="red", linetype="dashed", size=1) +
guides(fill = F) +
facet_grid(HV025~.) +
scale_fill_manual(values = colfunc(4)) +
theme_classic() 
La prevalencia de anemia en madres en una ciudad que sea capital se encuentra por encima de la mediana.
Prevalencia de anemia, edad y nivel educativo
dat %>%
group_by(HA1, HV109) %>%
summarise(an = mean(anem, na.rm = T)) %>%
ggplot(aes(y=an, x = HA1, fill = HV109, group = HV109)) +
geom_smooth() +
scale_fill_viridis_d(option = "cividis") +
scale_color_viridis_d(option = "cividis") +
labs( y = "Prevalencia de anemia", x = "Edad en años") +
facet_grid(.~HV109) +
guides(fill = F) +
theme_bw()
Las madres que han logrando alcanzar un nivel educativo superior tienen menor probabilidad de padecer de anemia a medida que pase el tiempo. Quizas esta reduccion se deba a que tienen mayor poder adquisitivo y pueden invertir mayor dinero en atenciones medicas, tanto curativas como preventiva, que las personas que no han alcanzado el nivel educativo superior.
Prevalencia de anemia, edad y embarazo
dat %>%
group_by(HA1, HA54) %>%
summarise(an = mean(anem, na.rm = T)) %>%
ggplot(aes(y=an, x = HA1, fill = HA54, col = HA54, group = HA54)) +
geom_smooth() +
scale_fill_viridis_d() +
scale_color_viridis_d() +
labs(fill = "Embarazada actualmente", col = "Embarazada actualmente", y = "Prevalencia de anemia", x = "Edad en años") +
theme_bw()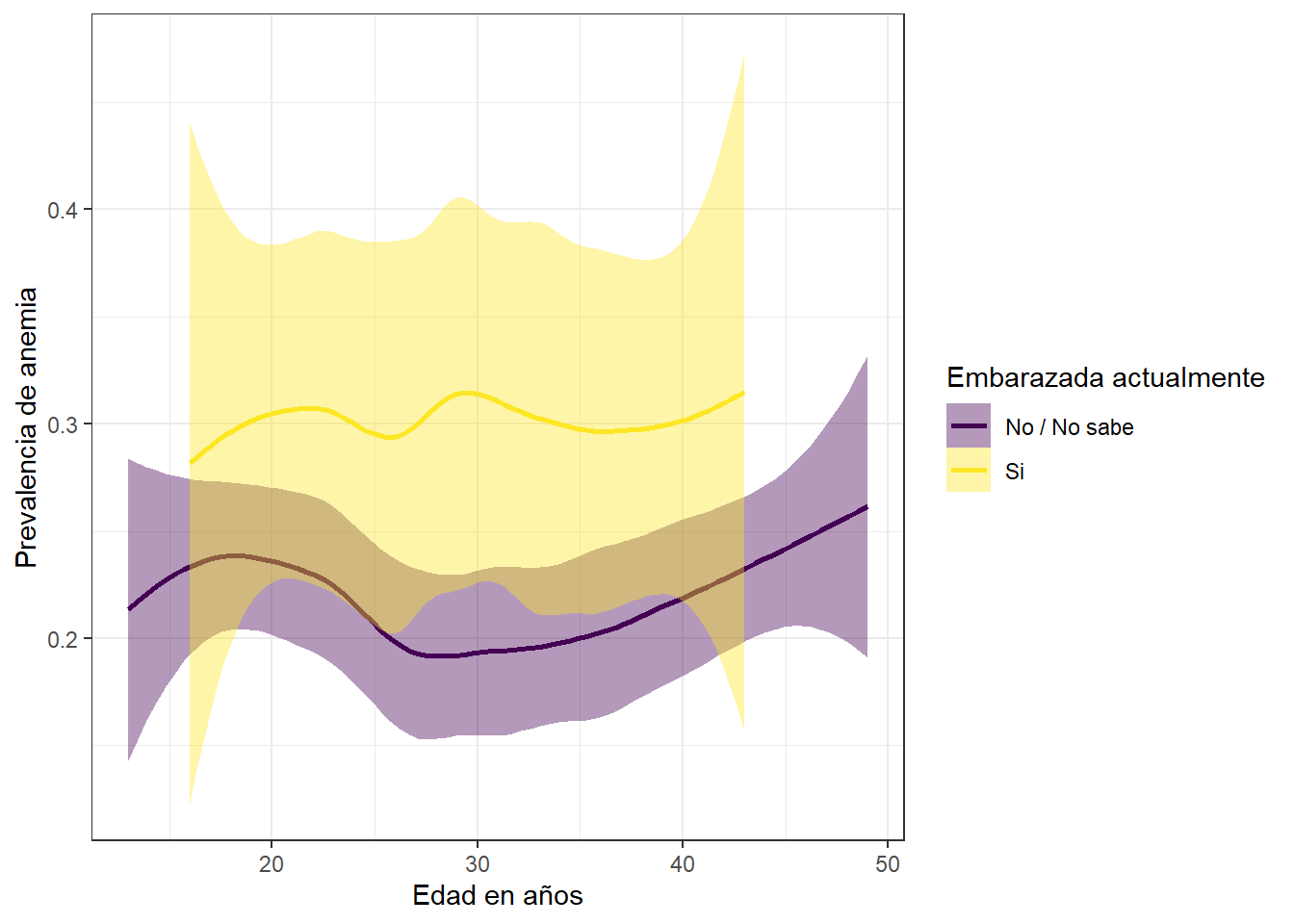
Es un hecho que las probabilidades de que las mujeres tengan anemia cuando estan embarazadas sean altas, y esto una vez mas se lgora afirmar mediante el grafico expuesto.
Prevalencia de anemia, edad e indice de riqueza
dat %>%
group_by(HA1, HV270) %>%
summarise(an = mean(anem, na.rm = T)) %>%
ggplot(aes(y=an, x = HA1, fill = HV270, group = HV270)) +
geom_smooth() +
scale_fill_viridis_d() +
scale_color_viridis_d() +
labs(fill = "Indice de riqueza", col = "Indice de riqueza", y = "Prevalencia de anemia", x = "Edad en años") +
facet_grid(.~HV270) +
guides(fill = F) +
theme_bw()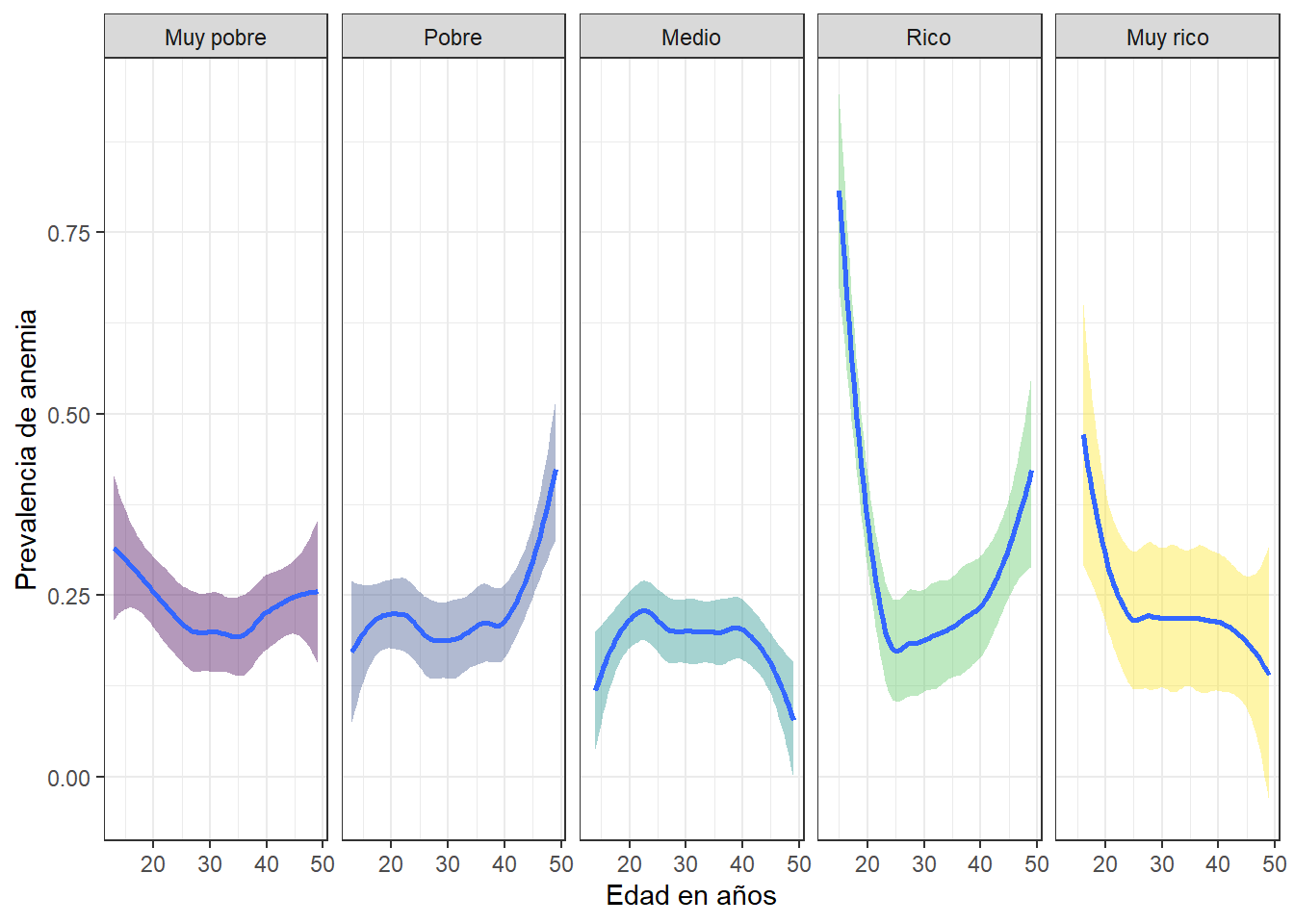
Durante las primeras etapas de vida, la prevalencia de anemia es mucho mayor en las mujeres con indices de riqueza rico y muy rico, pero con el paso del tiempo, este valor disminuira. Caso contrario sucede con las personas muy pobres y pobres, ya que, la prevalencia de anemia para esta poblacion se incrementa a medida que pasa el tiempo. La menor prevalencia de anemia la registra la poblacion correspondiente al indice de riqueza medio.
Prevalencia de y recepcion de hierro del MINSA
dat %>%
group_by(HA1, S465DB_A) %>%
summarise(an = mean(anem, na.rm = T)) %>%
filter(!is.na(S465DB_A)) %>%
ggplot(aes(y=an, x = HA1, fill = S465DB_A, col = S465DB_A, group = S465DB_A)) +
geom_smooth() +
scale_fill_viridis_d() +
scale_color_viridis_d() +
labs(fill = "Recibio hierro del MINSA", col = "Recibio hierro del MINSA", y = "Prevalencia de anemia", x = "Edad en años") +
facet_grid(.~S465DB_A) +
theme_bw() 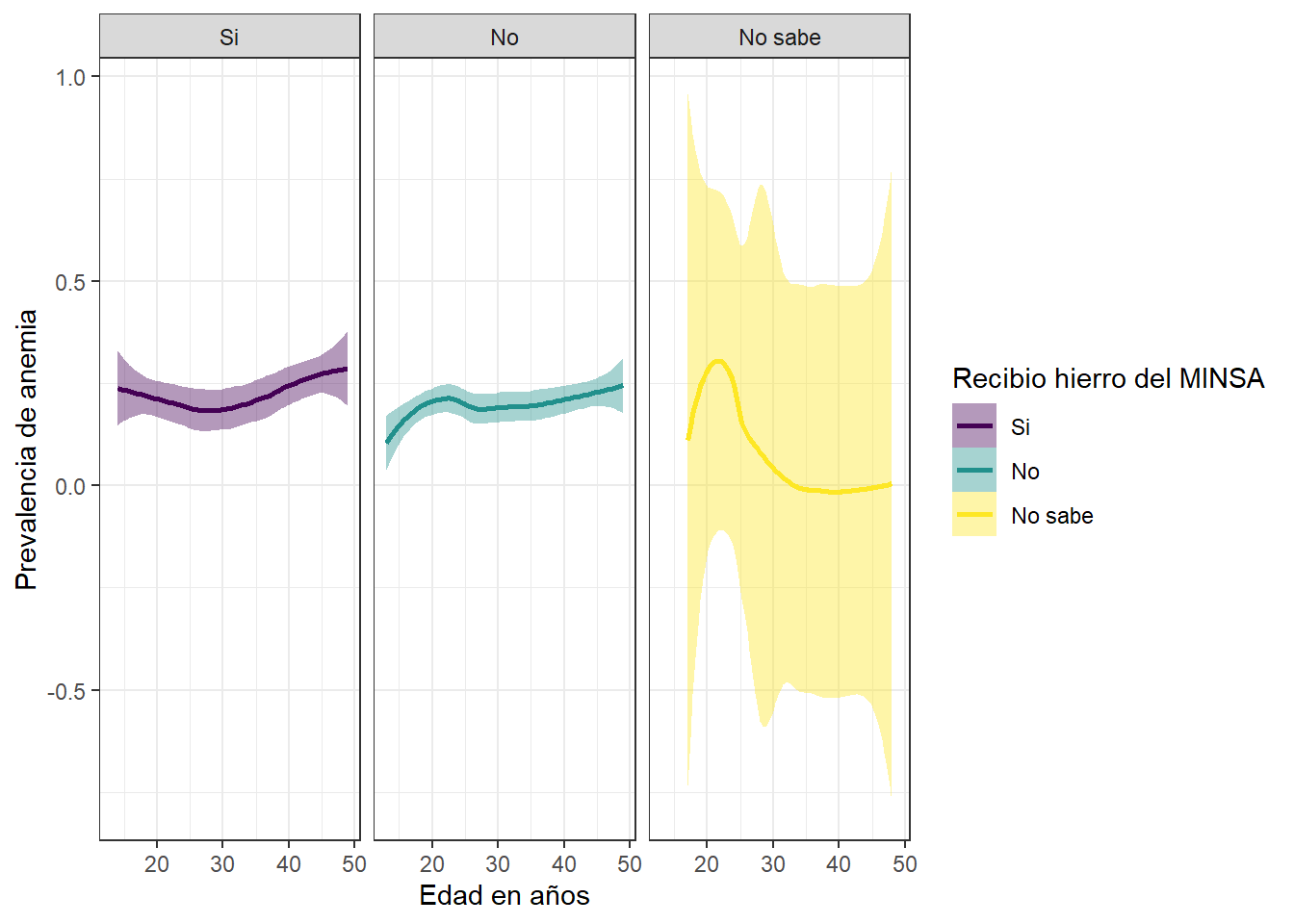
Es algo ilogico que las madres que SI RECIBIERON HIERRO DEL MINSA tengan mayor prevalencia de anemia que las madres que no lo hicieron. Ademas, las madres que no saben si recibieron hierro del MINSA son la poblacion que menor prevalencia de anemia tiene.
Prevalencia de anemia, edad y razones de no acudir a un centro de salud cuando el niño se enfermo
dat %>%
group_by(HA1, S470) %>%
summarise(an = mean(anem, na.rm = T)) %>%
filter(!is.na(S470)) %>%
ggplot(aes(y=an, x = HA1, fill = S470, col = S470, group = S470)) +
geom_smooth() +
scale_fill_viridis_d() +
scale_color_viridis_d() +
labs(fill = "Razones de no acudir al ES", col = "Razones de no acudir al ES", y = "Prevalencia de anemia", x = "Edad en años") +
facet_grid(.~S470) +
#guides(fill = F, col = F) +
theme_bw() +
theme(legend.position = "bottom") 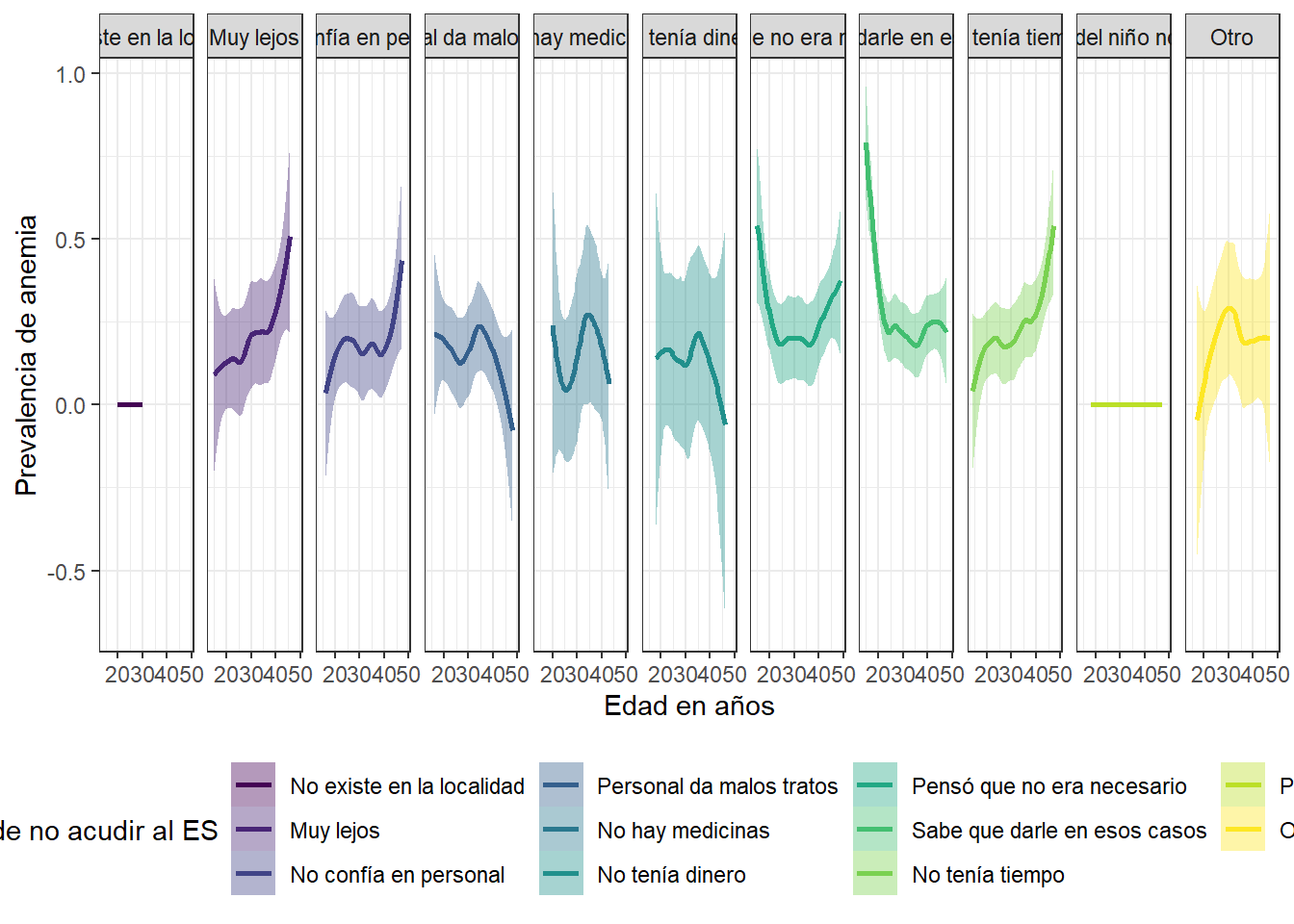
Las razones de no acudir al ES que hacen que la prevalencia de anemia aumente a medida que pasa el tiempo en las madres peruanas son las siguientes: el establecimiento de salud se encuentra muy lejos, las madres no confian en el personal y la poca disponibilidad de tiempo.
dat %>%
group_by(HA1, S470) %>%
summarise(an = mean(anem, na.rm = T)) %>%
filter(!is.na(S470)) %>%
ggplot(aes(y=an, x = HA1, fill = S470)) +
geom_violin() +
geom_jitter() +
geom_boxplot(fill = NA) +
geom_hline(aes(yintercept = median(an, na.rm = T)),
col = "red", linetype = "dashed", size = 1) +
scale_fill_viridis_d() +
scale_color_viridis_d() +
labs(fill = "Razones", col = "Razones", y = "Prevalencia de anemia", x = "Edad en años") +
facet_grid(.~S470) +
theme_bw() +
theme(legend.position = "bottom") 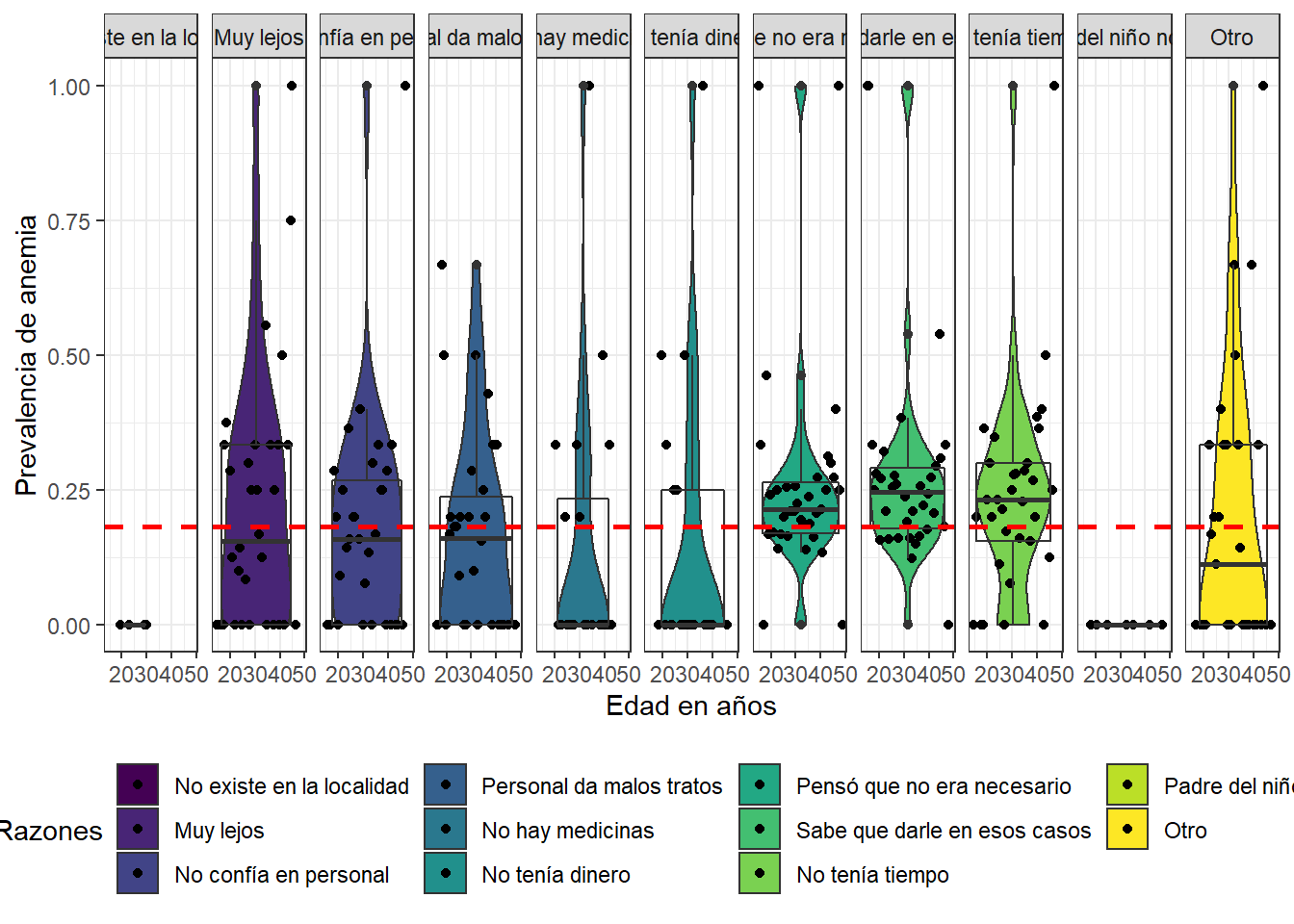
Prevalencia de anemia segun region y recepcion de hierro en jarabe MINSA
dat %>%
group_by(ubigeo, HV024, S465DB_A) %>%
summarise(an = mean(anem, na.rm = T)) %>%
filter(!is.na(S465DB_A)) %>%
ggplot(aes(y=an, x = HV024, fill = S465DB_A, group = S465DB_A)) +
geom_histogram(stat="identity", width=.5) +
scale_fill_viridis_d(option = "cividis") +
labs(x = "Region", y = "Prevalencia de anemia", fill = "Recibio hierro (jar) del MINSA") +
theme_bw() 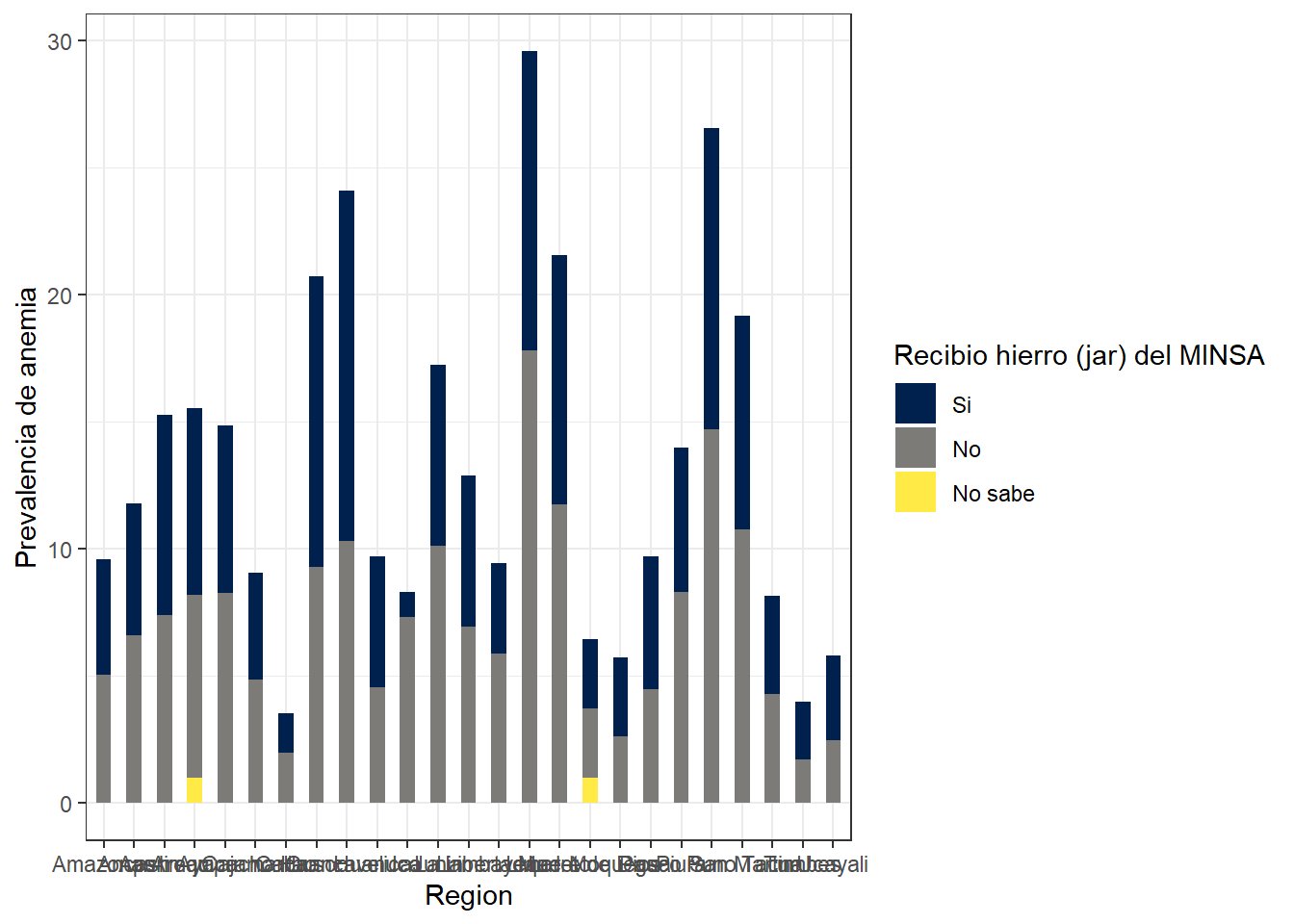
dat %>%
group_by(ubigeo, HV024, S465DB_A) %>%
summarise(an = mean(anem, na.rm = T)) %>%
filter(!is.na(S465DB_A)) %>%
ggplot(aes(x = HV024, y = an)) +
geom_violin(aes(fill = HV024)) +
geom_jitter(width = .2) +
geom_boxplot(fill = NA) +
labs(x = "Region", y = "Prevalencia de anemia") +
guides(fill = F) +
geom_hline(aes(yintercept=median(an,na.rm = T)),
color="red", linetype="dashed", size=1) +
facet_grid(S465DB_A~.) +
theme_classic() 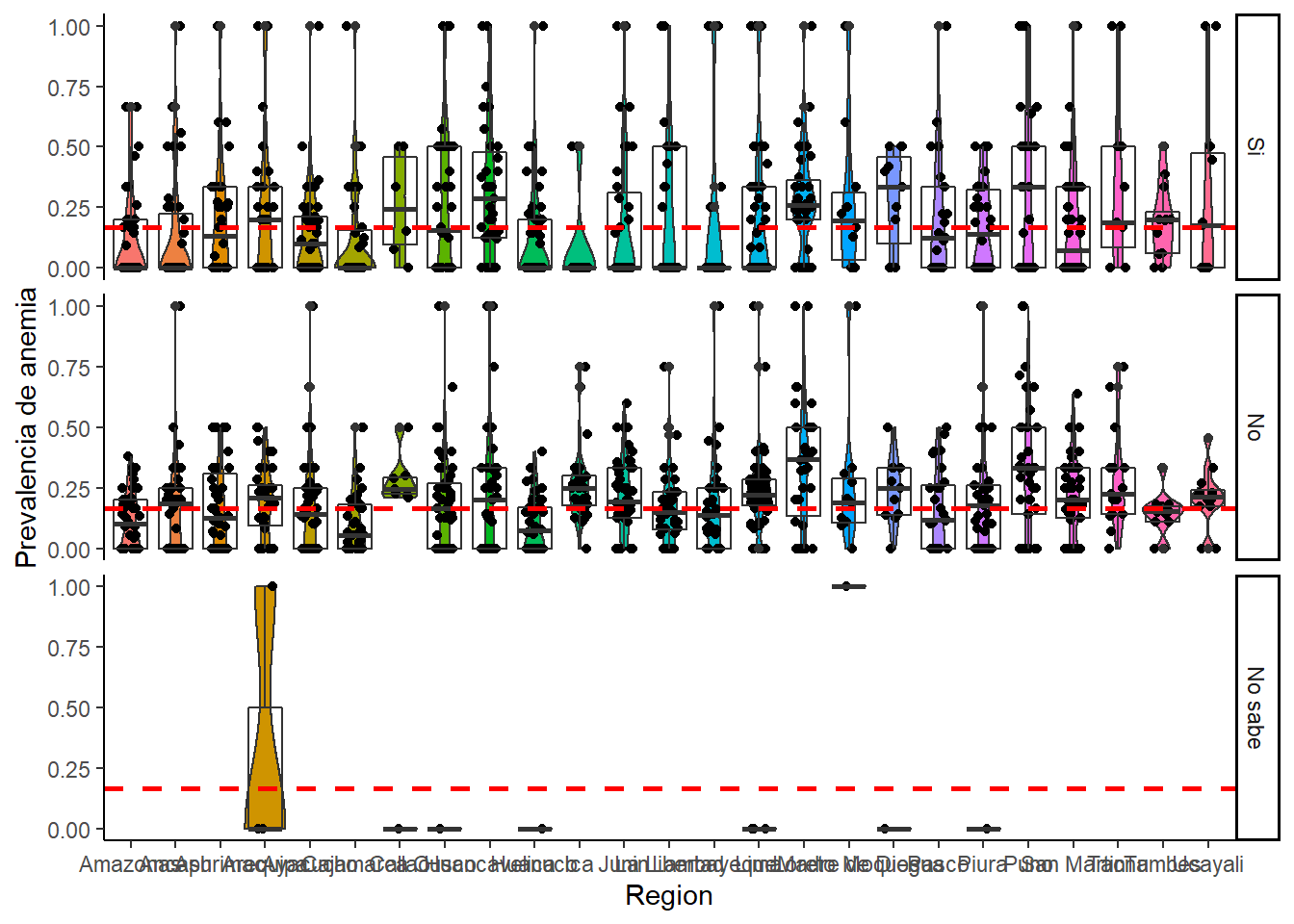
Prevalencia de anemia y edad segun posesion de radio en la vivienda
dat %>%
group_by(HA1, HV207) %>%
summarise(an = mean(anem, na.rm = T)) %>%
ggplot(aes(y = an, x = HA1, col = HV207, fill = HV207, group = HV207)) +
geom_smooth(alpha = .3) +
scale_fill_viridis_d() +
scale_color_viridis_d() +
labs(fill = "Tiene radio", col = "Tiene radio", y = "Prevalencia de anemia", x = "Edad en años") +
theme_bw()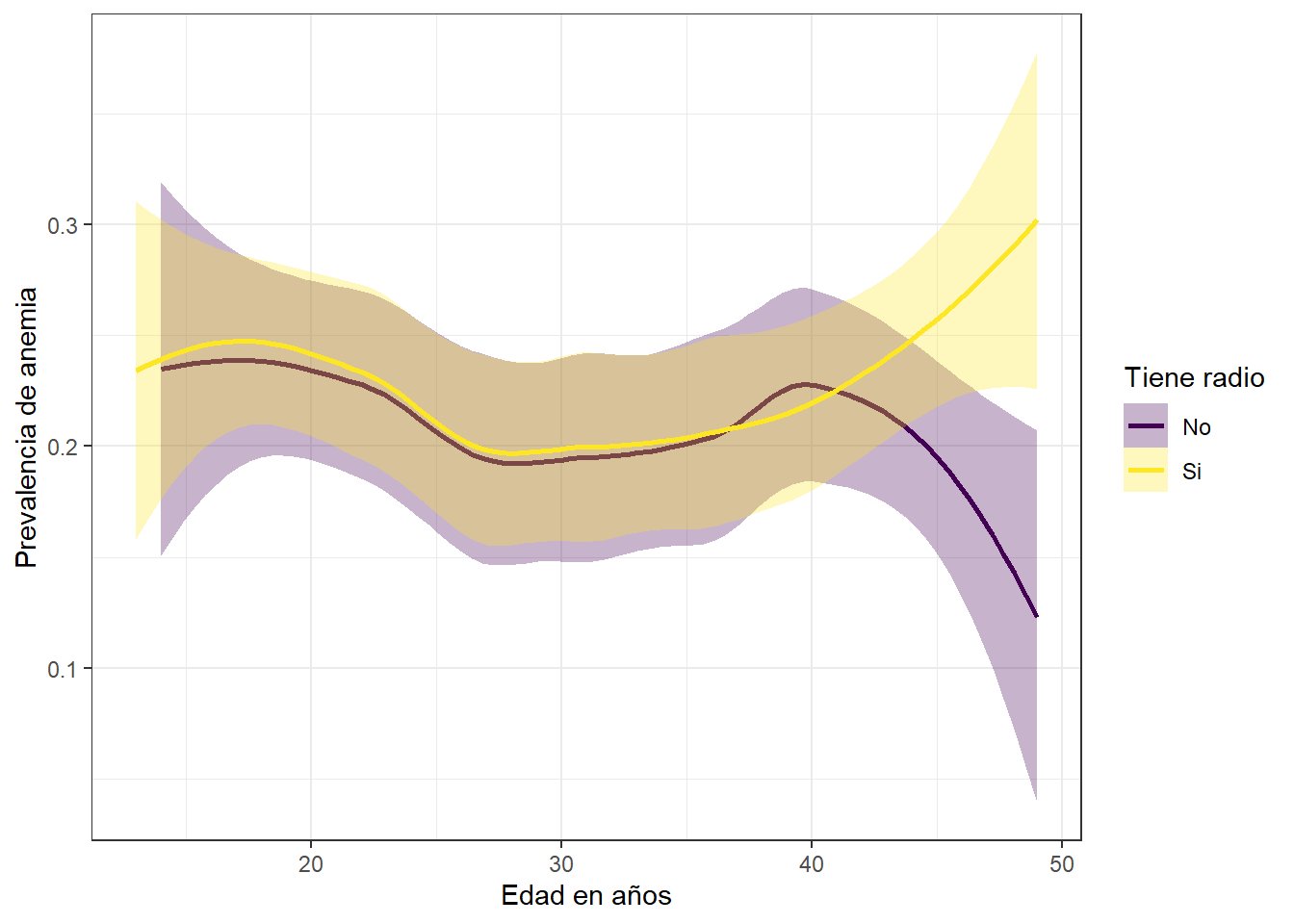
Las madres que tienen radio registran mayor prevalencia de anemia cuando se encuentran entre los 40 y 50 años.
Prevalencia de anemia y edad segun posesion de televisor en la vivienda
dat %>%
group_by(HA1, HV208) %>%
summarise(an = mean(anem, na.rm = T)) %>%
ggplot(aes(y = an, x = HA1, col = HV208, fill = HV208, group = HV208)) +
geom_smooth(alpha = .3) +
scale_fill_viridis_d() +
scale_color_viridis_d() +
labs(fill = "Tiene televisor", col = "Tiene televisor", y = "Prevalencia de anemia", x = "Edad en años") +
theme_bw()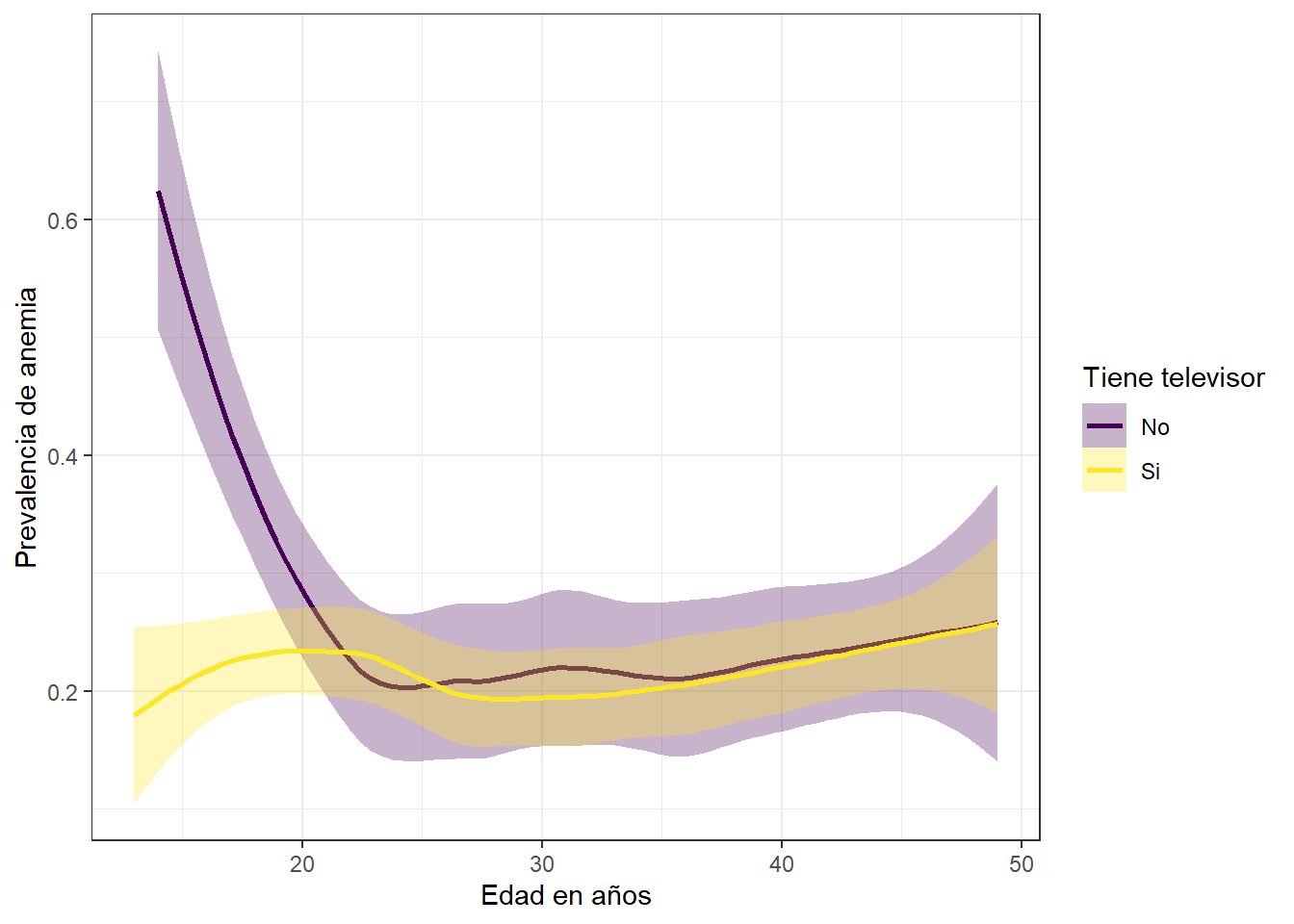
La tendencia de la prevalencia de anemia cuando comparada con la variable tiene televisor apunta a disminuir a medida que pasa el tiempo, independientemente de la posesion de un televisor en el hogar.
Prevalencia de anemia y edad segun el tipo de servicio higienico
dat %>%
group_by(HA1, HV225) %>%
summarise(an = mean(anem, na.rm = T)) %>%
filter(!is.na(HV225)) %>%
ggplot(aes(y = an, x = HA1, col = HV225, fill = HV225, group = HV225)) +
geom_smooth(alpha = .3) +
scale_fill_viridis_d() +
scale_color_viridis_d() +
labs(fill = "Comparte SS.HH", col = "Comparte SS.HH", y = "Prevalencia de anemia", x = "Edad en años") +
theme_bw()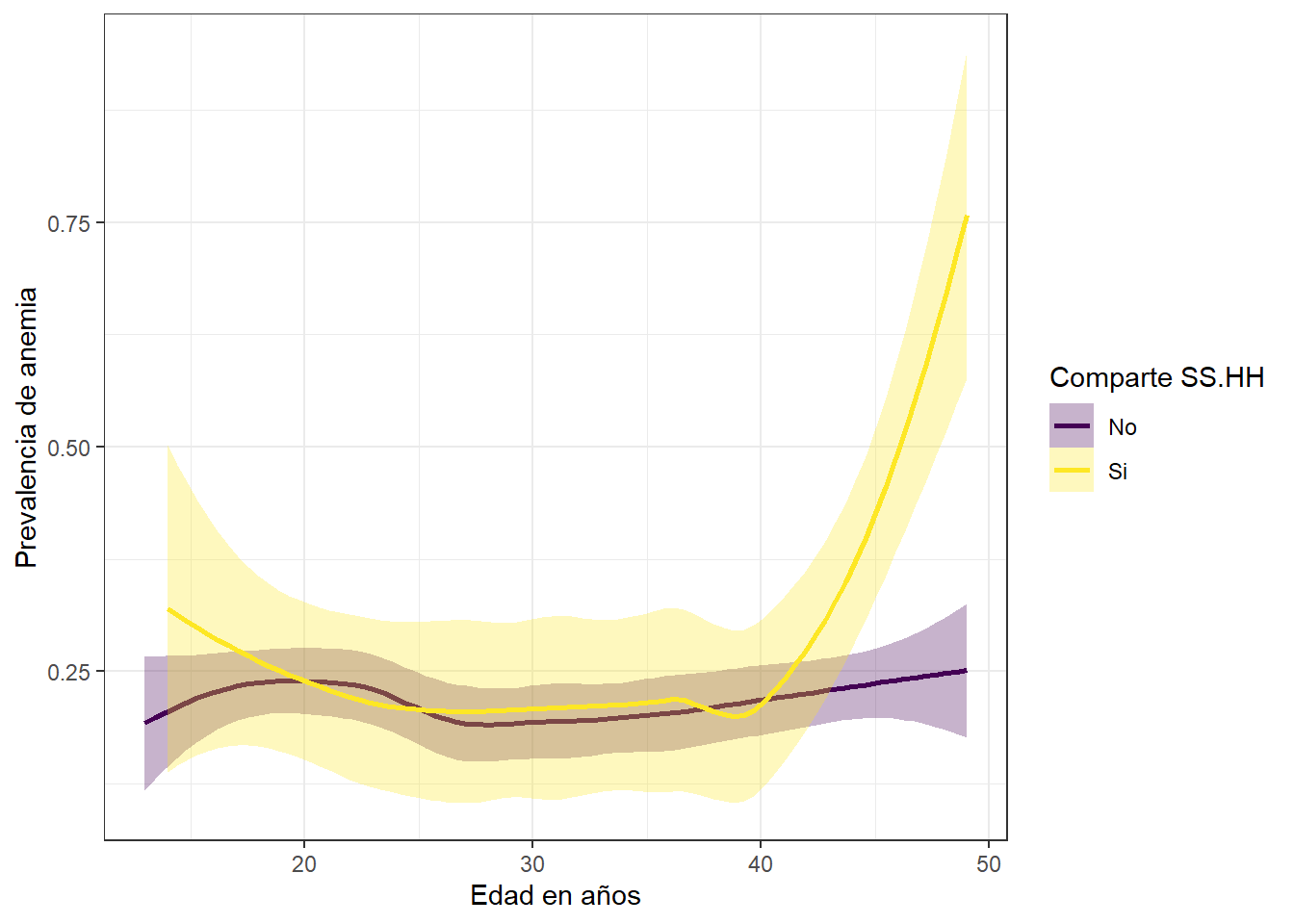
Aquellas madres que comparten los servicios higienicos tienen mayor probabilidad de padecer de anemia. Informacion proporcionada por la OMS afirma que la higiene y el saneamiento son factores causantes de esta enfermedad.
Prevalencia de anemia y edad segun la condicion de poseer cuarto para la cocina
dat %>%
group_by(HA1, HV242) %>%
summarise(an = mean(anem, na.rm = T)) %>%
filter(!is.na(HV242)) %>%
ggplot(aes(y = an, x = HA1, col = HV242, fill = HV242, group = HV242)) +
geom_smooth(alpha = .3) +
scale_fill_viridis_d() +
scale_color_viridis_d() +
labs(fill = "Tiene cuarto para la cocina", col = "Tiene cuarto para la cocina", y = "Prevalencia de anemia", x = "Edad en años") +
theme_bw()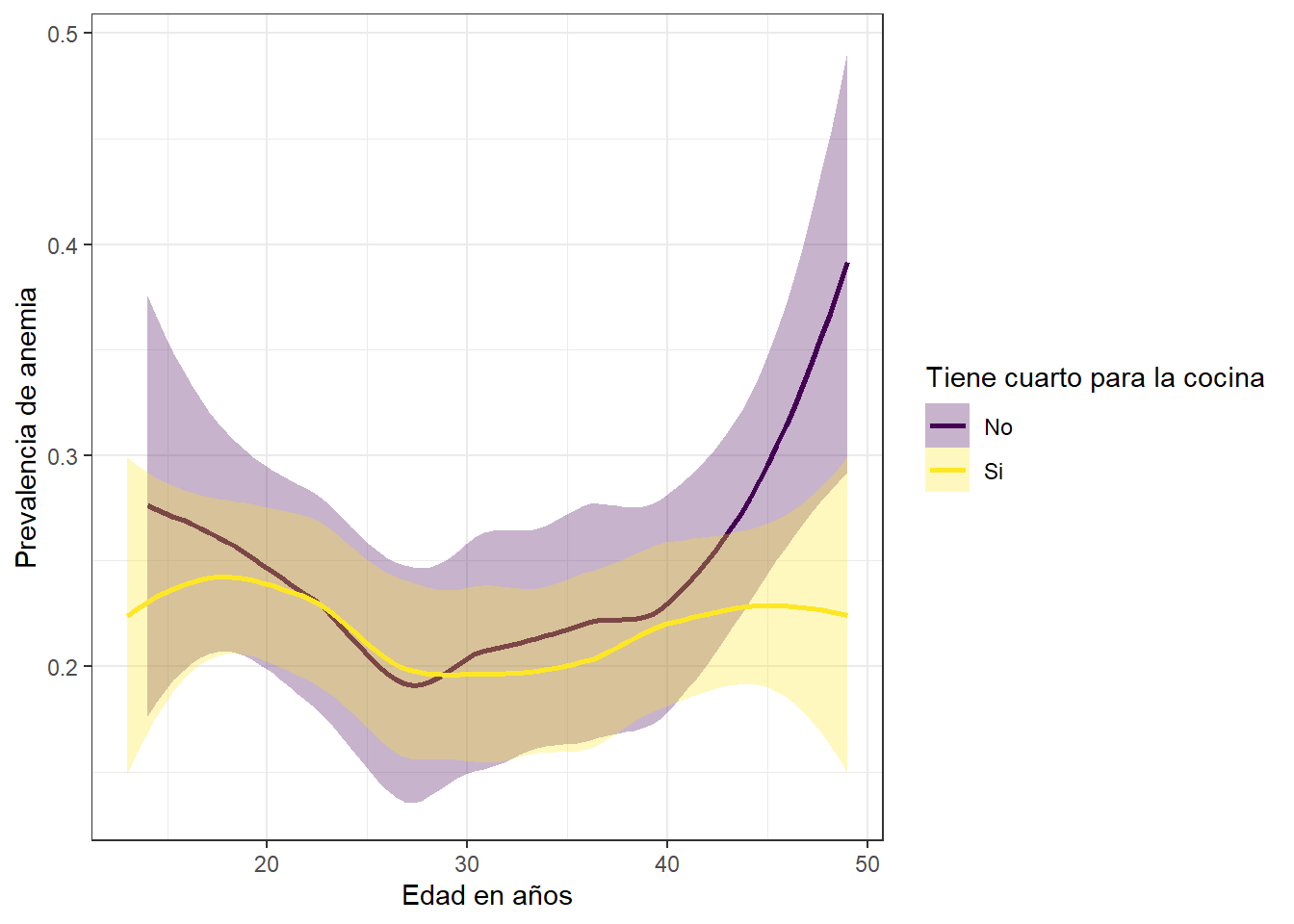
Las madres que no tienen un cuarto especifico para la cocina son aquellas que presentan mayor prevalencia de anemia en comparacion a las madres que si cuentan con uno.
Prevalencia de anemia, edad y disponibilidad de agua potable en el dia
dat %>%
group_by(HA1, SH42) %>%
summarise(an = mean(anem, na.rm = T)) %>%
filter(!is.na(SH42)) %>%
ggplot(aes(y = an, x = HA1, col = SH42, fill = SH42, group = SH42)) +
geom_smooth(alpha = .3) +
scale_fill_viridis_d() +
scale_color_viridis_d() +
labs(fill = "Agua potable todo el dia", col = "Agua potable todo el dia", y = "Prevalencia de anemia", x = "Edad en años") +
theme_bw()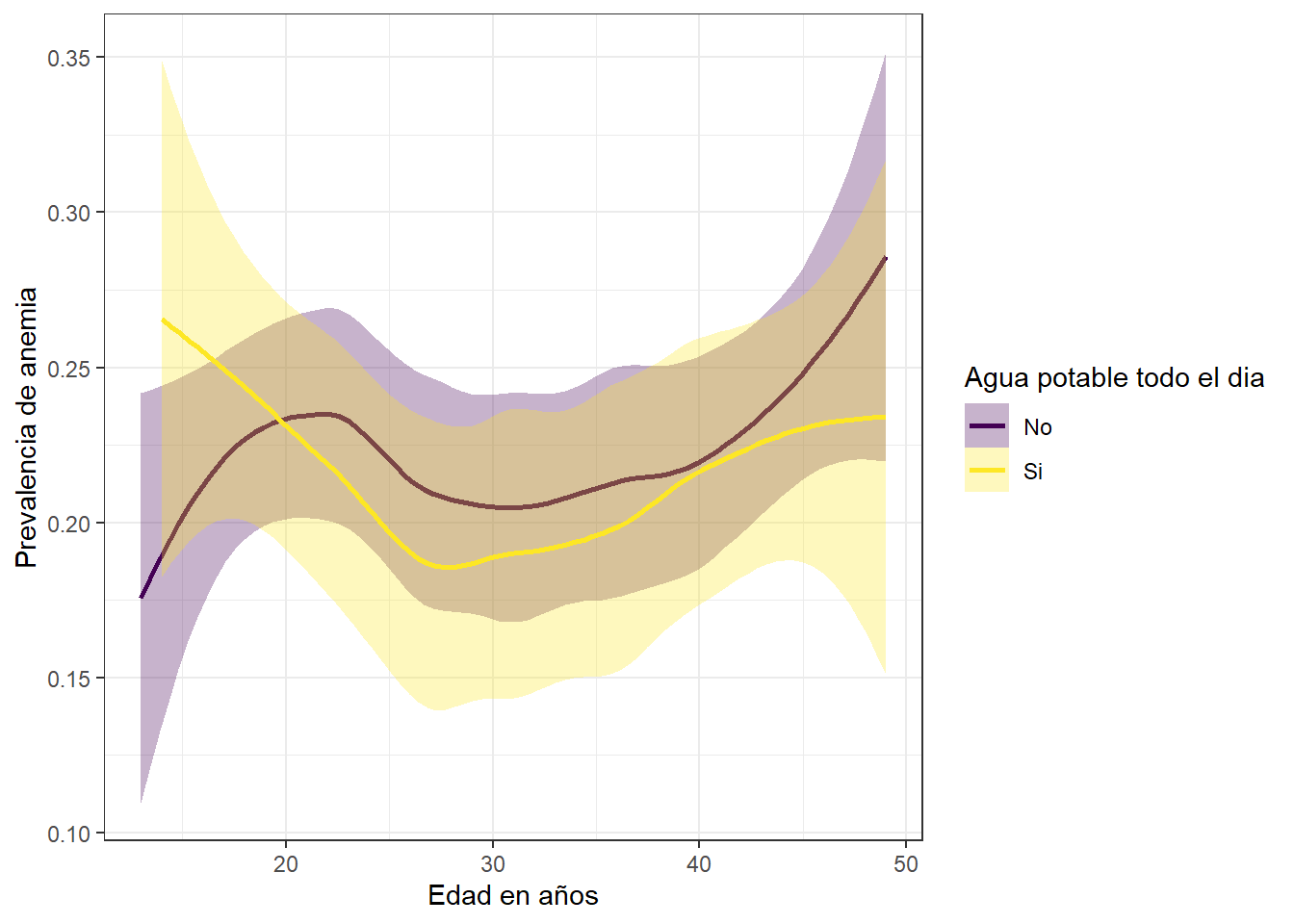
La prevalencia de anemia se registra con mayores valores en las madres que no disponen de agua potable todo el dia, la tendencia se incrementa a medida que pasa el tiempo.
Tendencia de anemia por region
dat %>%
group_by(HA53, HA2, SHREGION, HV024) %>%
summarise(an = mean(anem, na.rm = T)) %>%
filter(!is.na(HA2)) %>%
ggplot(aes(x = HA53, y = HA2, color = SHREGION)) +
geom_smooth(aes(group = SHREGION, col = SHREGION, fill = SHREGION),
method = "lm",
formula = y ~ log(x),
se = TRUE) + #muestra estimados de error
geom_point(shape = 1, size = 1) +
labs(fill = "REGION NATURAL", col = "REGION NATURAL", x = "Hemoglobina en sangre (g-dl - 1 decimal)",
y = "Peso en KG" ) +
theme_bw()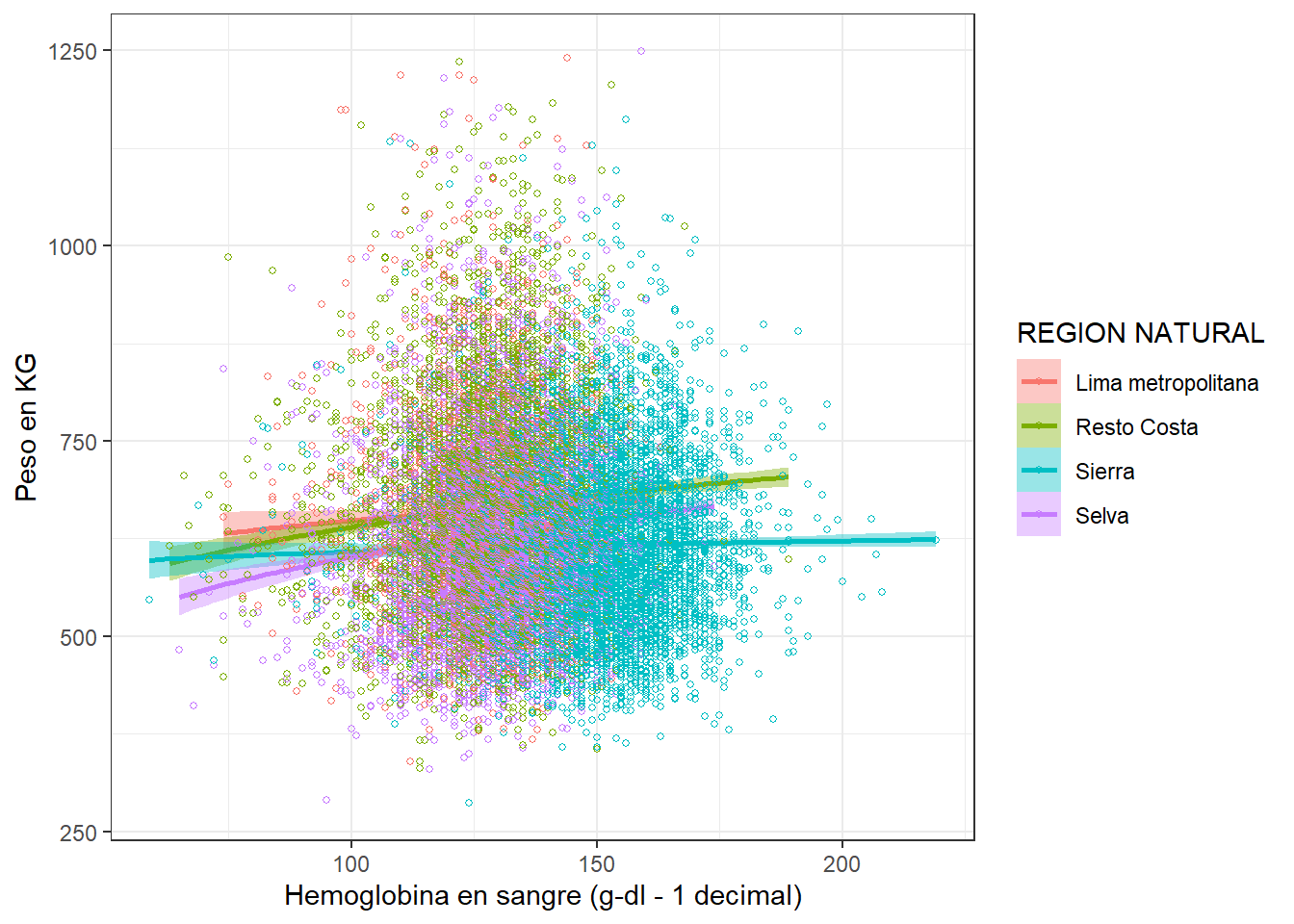
2.4.1 Variables nulas
En esta seccion, se presentaran todas las variables que por alguna razon no albergan datos en la E
Prevalencia de anemia y edad segun la condicion de practicar lactancia materna
dat %>%
group_by(HV104, HA1, S466GA) %>%
summarise(an = mean(anem, na.rm = T)) %>%
ggplot(aes(y=an, x = HA1, fill = HV104, col = HV104, group = HV104)) +
geom_point(alpha = .3) +
geom_smooth() +
scale_fill_viridis_d() +
scale_color_viridis_d() +
labs(fill = "Sex", col = "Sex", y = "Anemia prevalence", x = "Age in years") +
facet_grid(.~S466GA) +
theme_bw() 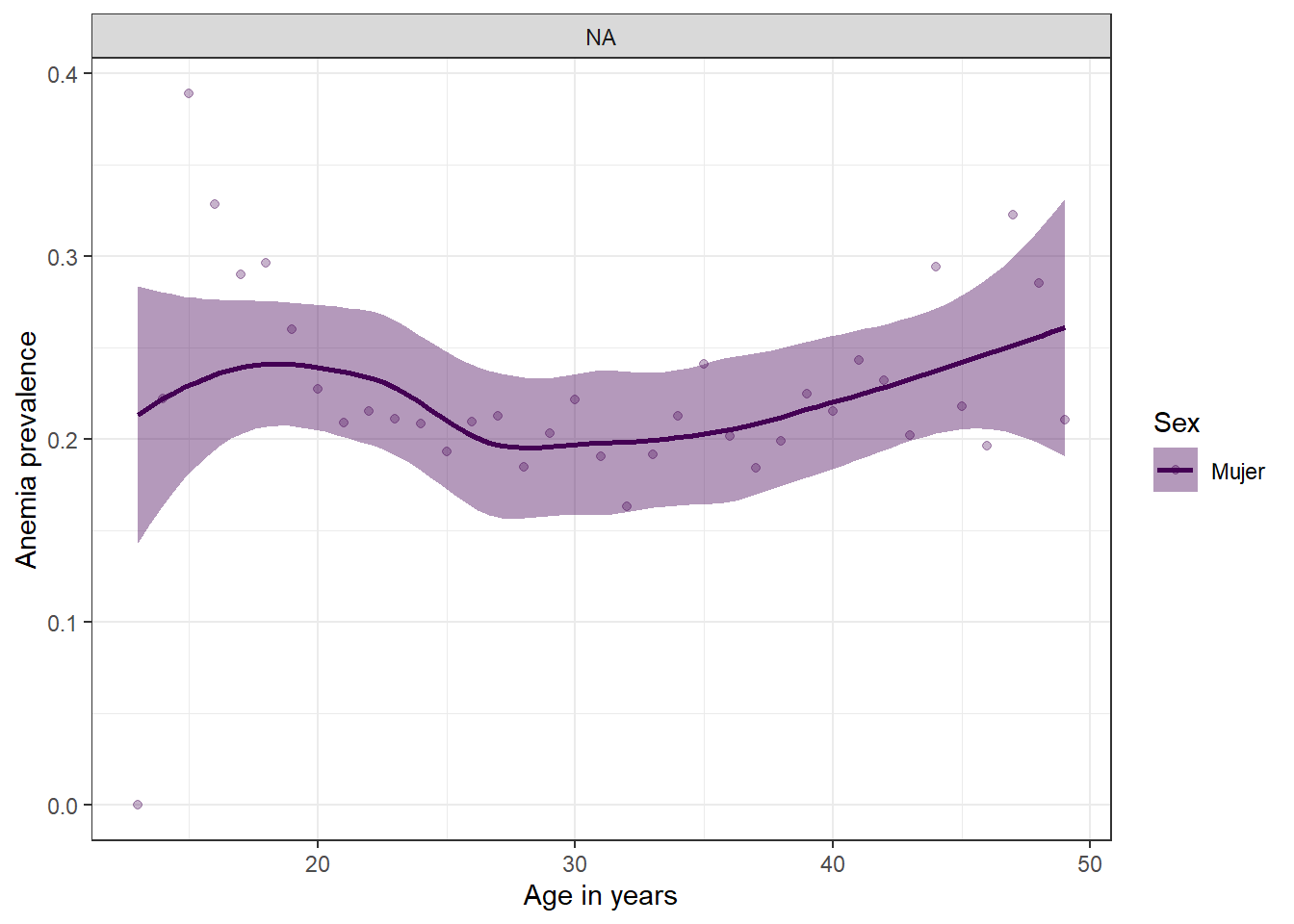
El grafico muestra las variables definidas previamente en la estetica de ggplot (eje x e y), pero no se pudo hacer el facet segun la variable S466GA “practico como dar de lactar al niño/a”, debido a que la variable no registra datos. Esto lo podemos comprobar de la siguiente manera:
## [1] 19669El resultado de la suma es 19669, que es el numero total de observaciones dentro de la base de datos.
dat %>%
group_by(HV104, HA1, S466GB) %>%
summarise(an = mean(anem, na.rm = T)) %>%
ggplot(aes(y=an, x = HA1, fill = HV104, col = HV104, group = HV104)) +
geom_point(alpha = .3) +
geom_smooth() +
scale_fill_viridis_d() +
scale_color_viridis_d() +
labs(fill = "Sex", col = "Sex", y = "Anemia prevalence", x = "Age in years") +
facet_grid(.~S466GB) +
theme_bw() 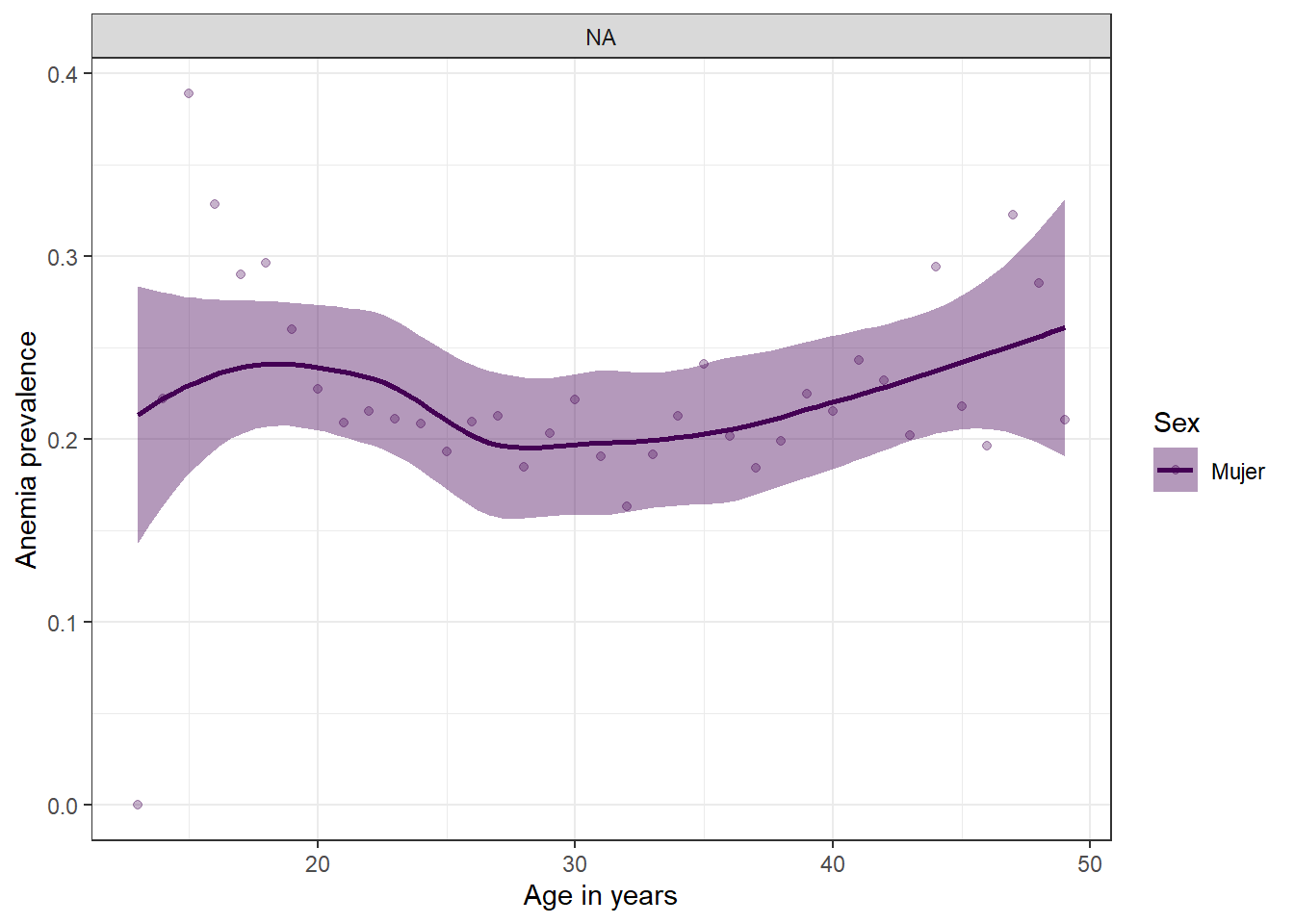
El grafico muestra las variables definidas previamente en la estetica de ggplot (eje x e y), pero no se pudo hacer el facet segun la variable S466GB “practico como preparar los alimentos al niño/a”, debido a que la variable no registra datos. Esto lo podemos comprobar de la siguiente manera:
## [1] 19669El resultado de la suma es 19669, que es el numero total de observaciones dentro de la base de datos.
Prevalencia de anemia y edad según la ubicacion de la fuente de agua
dat %>%
group_by(HA1, HV235) %>%
summarise(an = mean(anem, na.rm = T)) %>%
ggplot(aes(y = an, x = HA1)) +
geom_smooth(alpha = .3) +
scale_fill_viridis_d() +
scale_color_viridis_d() +
facet_grid(.~HV235) +
labs(fill = "Fuente de agua", y = "Prevalencia de anemia", x = "Edad en años") +
theme_bw()
El grafico muestra las variables definidas previamente en la estetica de ggplot (eje x e y), pero no se pudo hacer el facet segun la variable HV235 “ubicacion de la fuente de agua”, debido a que la variable no registra datos. Esto lo podemos comprobar de la siguiente manera:
## [1] 19669El resultado de la suma es 19669, que es el numero total de observaciones dentro de la base de datos.
Prevalencia de anemia y edad segun el lugar en el que se prepara la comida
dat %>%
group_by(HA1, HV241) %>%
summarise(an = mean(anem, na.rm = T)) %>%
ggplot(aes(y = an, x = HA1)) +
geom_smooth(alpha = .3) +
scale_fill_viridis_d() +
scale_color_viridis_d() +
facet_grid(.~HV241) +
labs(fill = "Lugar donde se prepara la comida", col = "Lugar donde se prepara la comida", y = "Prevalencia de anemia", x = "Edad en años") +
theme_bw()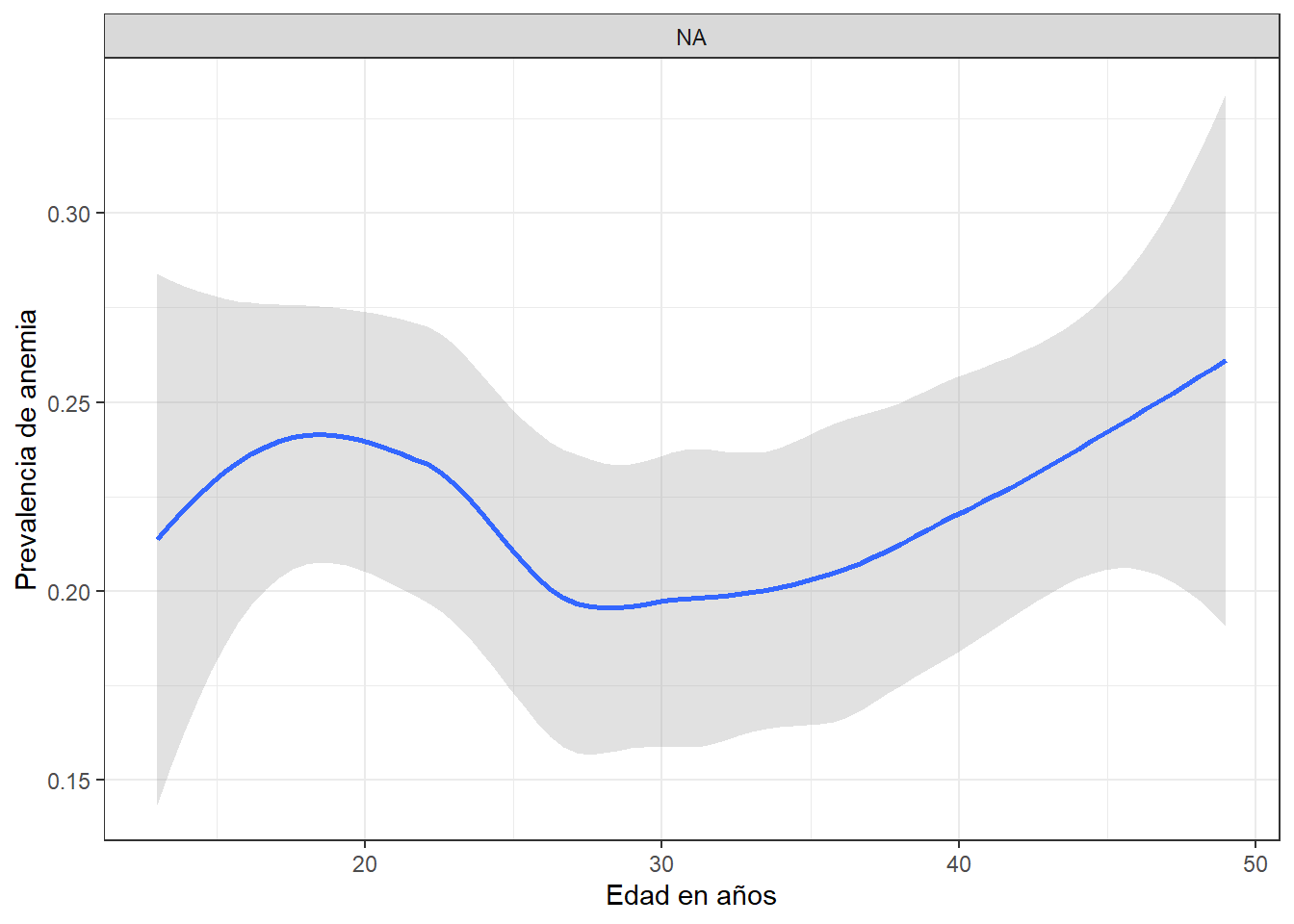
El grafico muestra las variables definidas previamente en la estetica de ggplot (eje x e y), pero no se pudo hacer el facet segun la variable HV241 “Comida preparada en el hogar, edificio separado o fuera”, debido a que la variable no registra datos. Esto lo podemos comprobar de la siguiente manera:
## [1] 196692.5 Conclusiones
La anemia es una enfermedad que se explica por un bajo nivel de hemoglobina en sangre y que trae consigo problemas al momento de transportar el oxigeno a las celulas del cuerpo. Existen diversos factores causantes de esta enfermedad, entre ellos tenemos a factores nutricionales, factores socioeconomicos, factores culturales, etc. Es por ello que, las autoridades deben tomar medidas multisectoriales para poder afrontar esta enfermedad que sigue siendo un problema para los peruanos.
Para el año 2018, el 21.2% de las madres peruanas tuvieron esta enfermedad. Existen categorias que clasifican a las madres segun el nivel de anemia que tienen. Anemia severa (0.1%), anemia moderada (2.3%), y anemia leve (18.8%).
Dentro de los factores asociados a causar una mayor o menor prevalencia de anemia en las madres peruanas tenemos al tipo de ciudad (mayor prevalencia en la capital y menor prevalencia en el ciudades pequeñas), el tipo de residencia (madres en el area rural tienen mayor prevalencia de anemia), la edad (madres entre los 40 y 50 años registran mayor prevalencia de anemia), el embarazo (madres embarazadas tienen mayor prevalencia de anemia), el indice de riqueza (Durante las primeras etapas de vida, la prevalencia de anemia es mucho mayor en las mujeres con indices de riqueza rico y muy rico, pero disminuira con el paso del tiempo, las mujeres con indices de riqueza muy y pobre tienen menor prevalencia de anemia que la otra poblacion mencionada, pero la prevalencia aumentara a medida que pase el tiempo).
2.6 Decision Trees
Las variables que no se tomaran en cuenta por no registrar ningun dato son las siguientes:HV241, HV235, S466GB, S466GA
library(rpart.plot)
library(rpart)
library(tidyverse)
library(caret)
library(caTools)
dat_non_na <- dat %>%
filter_at(vars(SH42,HV242, HV225, S465DB_A, S470), all_vars(!is.na(.))) %>%
select(-HV241, -HV235, -S466GB, -S466GA, -HA57, -anem, -ubigeo) #quite ubigeo2.6.1 Creacion de sets de entreamiento y prueba
Despues de haber dejado limpia la base que utilizaremos para el modelo, necesitamos particionarla en dos subconjuntos. Uno destinado al entrenamiento y otro a la prueba. La distribucion que se utilizara para la particion de los datasets sera 80% para el entrenamiento y 20% para la prueba.
set.seed(1234) #para hacer reproducible el ejemplo
split <- sample.split(dat_non_na$Anemic, SplitRatio = 0.80)
dat_train <- subset(dat_non_na, split == TRUE)
dat_test <- subset(dat_non_na, split == FALSE)Ahora verificamos si es que la distribucion de la variable Anemic es la misma en ambas bases de datos.
##
## No Anemic Anemic
## 1777 462##
## No Anemic Anemic
## 444 116La relacion No Ameic/Anemic se encuentra alrededor de 3.8 en ambas bases.
2.6.2 Entrenando el modelo/ Cross Validation
La variable que es de nuestro interes para la clasificacion es Anemic. Se entrenara el modelo sobre k = 10 subconjuntos de datos.

A continuacion se desarrollara el modelo sobre k = 10
cvtree1 <- lapply(folds, function(x){
training_fold <- dat_train[-x,]
test_fold <- dat_train[x, ]
clasificador <- rpart(formula = Anemic ~ .,data = dat_train)
pred <- predict(clasificador, newdata = test_fold, type = "class")
cm <- table(test_fold$Anemic, pred)
precision <- (cm[1,1]+cm[2,2]) / (cm[1,1] + cm[2,2] +cm[1,2] + cm[2,1])
return(precision)
})
precisiontree1 <- mean(as.numeric(cvtree1))
precisiontree1 ## [1] 0.96024952.6.3 Podando el arbol
##
## Classification tree:
## rpart(formula = Anemic ~ ., data = dat_train)
##
## Variables actually used in tree construction:
## [1] HA53 HA54 HV024 SHREGION
##
## Root node error: 462/2239 = 0.20634
##
## n= 2239
##
## CP nsplit rel error xerror xstd
## 1 0.668831 0 1.00000 1.00000 0.041447
## 2 0.028139 1 0.33117 0.33117 0.025842
## 3 0.010823 5 0.21429 0.21429 0.021055
## 4 0.010000 7 0.19264 0.19913 0.020330El xerror con menor valor para el arbol de decision que hemos creado es 0.21645. Por tal motivo, utilizaremos el CP que le corresponde a ese valor y es 0.01
