5 Machine Learning - Classificação
A Classificação é uma área do Machine Learning que como o próprio nome diz classifica em classes as observações a partir de variáveis preditivas, por exemplo, em uma classe binária, as observações serão classificadas em 0 ou 1.
5.1 Base de dados
A base de dados utilizada para aplicação dos algoritmos de classificação são de observações obtidos no seguinte endereço:
As amostras são de pacientes do Dr.William H.Wolberg do Hospitais da Universidadede Wisconsin, Madison, Wisconsin, EUA. As amostras foram coletadas periodicamente conforme os casos ocorriam. O banco de dados possui um total de 699 observações e 11 variáveis, destas variáveis foi excluída a variável de identificação do paciente. As variaveis são descritas adiante, onde a variável classe será a variável que queremos prevê, e as demais são as variáveis explicativas, conforme é mostrado a seguir:
- Espessura de aglomerado 1-10
- Uniformidade do tamanhoda célula 1-10
- Uniformidade da forma celular 1-10
- Adesão marginal 1-10
- Tamanho de célula epitelial única 1-10
- Núcleos nus 1-10
- Cromatina Suave 1-10
- Nucléolos normais 1-10
- Mitoses 1-10
- Classe (2 para benigno, 4 para maligno)
Os algoritmos de classificação serão treinados e posteriormente será submetida a base de dados de teste para verificar o desempenho dos algoritmos. Deseja-se prever o diagnostico do paciente, sendo 2 para a classe benigna e 4 para a classe maligna, conforme segue:
2: Não Câncer (Tumor benigno);
4: Câncer (Tumor maligno).
5.2 Análise exploratória
Antes de seguir com a abordagem e aplicação dos modelos de Machine Learning é imprescindível que seja realizada uma análise exploratória dos dados.
5.2.1 Preparação dos dados
Comece carregando o banco de dados, onde header = TRUE significa que a primeira linha do arquivo deve ser interpretada como os nomes das colunas e sep = indica o tipo de separador de colunas.
dados = read.csv("Cancer_Wisconsin.csv", header = T, sep = ";")5.2.2 Pacotes utilizados
Carregando os pacotes que serão utilizados ao longo da análise, são eles, tidymodels para uso dos algoritmos de Machine Learning, corrplot e Hmisc para verificar a correlação, knitr e kableExtra para geração de tabelas.
library(tidymodels)
library(corrplot)
library(Hmisc)
library(knitr)
library(kableExtra)Para visualizar as primeiras linhas dos dados utiliza-se o comando a seguir.
dados %>% head() O pipe (%>%) usa o valor resultante do lado esquerdo como primeiro argumento da função do lado direito, conhecido também como operador de encadeamento. A função glimpse() retorna as primeiras observações e o tipo das variáveis.
glimpse(dados)## Rows: 699
## Columns: 11
## $ Nº.Ident <int> 1000025, 1002945, 1015425, 1016277, 1017023, 1017…
## $ Espessura_aglomerado <int> 5, 5, 3, 6, 4, 8, 1, 2, 2, 4, 1, 2, 5, 1, 8, 7, 4…
## $ Uni.tamanho_celula <int> 1, 4, 1, 8, 1, 10, 1, 1, 1, 2, 1, 1, 3, 1, 7, 4, …
## $ Uni.forma_celular <int> 1, 4, 1, 8, 1, 10, 1, 2, 1, 1, 1, 1, 3, 1, 5, 6, …
## $ Adesao_marginal <int> 1, 5, 1, 1, 3, 8, 1, 1, 1, 1, 1, 1, 3, 1, 10, 4, …
## $ T.celula_epitelial <int> 2, 7, 2, 3, 2, 7, 2, 2, 2, 2, 1, 2, 2, 2, 7, 6, 2…
## $ nucleos_nus <int> 1, 10, 2, 4, 1, 10, 10, 1, 1, 1, 1, 1, 3, 3, 9, 1…
## $ cromatina_suave <int> 3, 3, 3, 3, 3, 9, 3, 3, 1, 2, 3, 2, 4, 3, 5, 4, 2…
## $ nucleolos_normais <int> 1, 2, 1, 7, 1, 7, 1, 1, 1, 1, 1, 1, 4, 1, 5, 3, 1…
## $ mitoses <int> 1, 1, 1, 1, 1, 1, 1, 1, 5, 1, 1, 1, 1, 1, 4, 1, 1…
## $ classe <int> 2, 2, 2, 2, 2, 4, 2, 2, 2, 2, 2, 2, 4, 2, 4, 4, 2…A primeira coluna dos dados não é necessária para a análise, pode-se exclui-lá do conjunto de dados.
dados$Nº.Ident = NULLA variável resposta classe precisa ser transformada em fator, utilizando o comando as_factor obtêm-se essa transformação.
dados$classe = factor(dados$classe)Após transformar a variável classe em fator pode-se confirmar a alteração executando novamente o comando abaixo.
glimpse(dados)## Rows: 699
## Columns: 10
## $ Espessura_aglomerado <int> 5, 5, 3, 6, 4, 8, 1, 2, 2, 4, 1, 2, 5, 1, 8, 7, 4…
## $ Uni.tamanho_celula <int> 1, 4, 1, 8, 1, 10, 1, 1, 1, 2, 1, 1, 3, 1, 7, 4, …
## $ Uni.forma_celular <int> 1, 4, 1, 8, 1, 10, 1, 2, 1, 1, 1, 1, 3, 1, 5, 6, …
## $ Adesao_marginal <int> 1, 5, 1, 1, 3, 8, 1, 1, 1, 1, 1, 1, 3, 1, 10, 4, …
## $ T.celula_epitelial <int> 2, 7, 2, 3, 2, 7, 2, 2, 2, 2, 1, 2, 2, 2, 7, 6, 2…
## $ nucleos_nus <int> 1, 10, 2, 4, 1, 10, 10, 1, 1, 1, 1, 1, 3, 3, 9, 1…
## $ cromatina_suave <int> 3, 3, 3, 3, 3, 9, 3, 3, 1, 2, 3, 2, 4, 3, 5, 4, 2…
## $ nucleolos_normais <int> 1, 2, 1, 7, 1, 7, 1, 1, 1, 1, 1, 1, 4, 1, 5, 3, 1…
## $ mitoses <int> 1, 1, 1, 1, 1, 1, 1, 1, 5, 1, 1, 1, 1, 1, 4, 1, 1…
## $ classe <fct> 2, 2, 2, 2, 2, 4, 2, 2, 2, 2, 2, 2, 4, 2, 4, 4, 2…Uma maneira de verificar a existência de dados faltantes é usando a função complete.cases() que retorna os casos completos e a exclamação ! funciona como o operador de negação, que retorna o contrário do que foi solicitado, neste caso é retornado as observações faltantes.
sum(!complete.cases(dados))## [1] 16No banco de dados é observado um total de 16 observações faltantes. Essa observações faltantes serão tratadas adiante utilizando o pacote recipes do tidymodels, pois este processo só poderá ser aplicado aos dados de treinamento.
Pode-se obter algumas medidas descritivas usando a função summary
summary(dados)## Espessura_aglomerado Uni.tamanho_celula Uni.forma_celular Adesao_marginal
## Min. : 1.000 Min. : 1.000 Min. : 1.000 Min. : 1.000
## 1st Qu.: 2.000 1st Qu.: 1.000 1st Qu.: 1.000 1st Qu.: 1.000
## Median : 4.000 Median : 1.000 Median : 1.000 Median : 1.000
## Mean : 4.418 Mean : 3.134 Mean : 3.207 Mean : 2.807
## 3rd Qu.: 6.000 3rd Qu.: 5.000 3rd Qu.: 5.000 3rd Qu.: 4.000
## Max. :10.000 Max. :10.000 Max. :10.000 Max. :10.000
##
## T.celula_epitelial nucleos_nus cromatina_suave nucleolos_normais
## Min. : 1.000 Min. : 1.000 Min. : 1.000 Min. : 1.000
## 1st Qu.: 2.000 1st Qu.: 1.000 1st Qu.: 2.000 1st Qu.: 1.000
## Median : 2.000 Median : 1.000 Median : 3.000 Median : 1.000
## Mean : 3.216 Mean : 3.545 Mean : 3.438 Mean : 2.867
## 3rd Qu.: 4.000 3rd Qu.: 6.000 3rd Qu.: 5.000 3rd Qu.: 4.000
## Max. :10.000 Max. :10.000 Max. :10.000 Max. :10.000
## NA's :16
## mitoses classe
## Min. : 1.000 2:458
## 1st Qu.: 1.000 4:241
## Median : 1.000
## Mean : 1.589
## 3rd Qu.: 1.000
## Max. :10.000
## Observa-se que a média retornada não é um número inteiro, pois os dados estão em uma escala ordinal de 1 a 10, portanto a mediana será utilizada para a imputação dos valores faltantes, além disso, podemos notar pelo o conjunto de gráficos na Figura 5.1 que os dados não são simétricos.
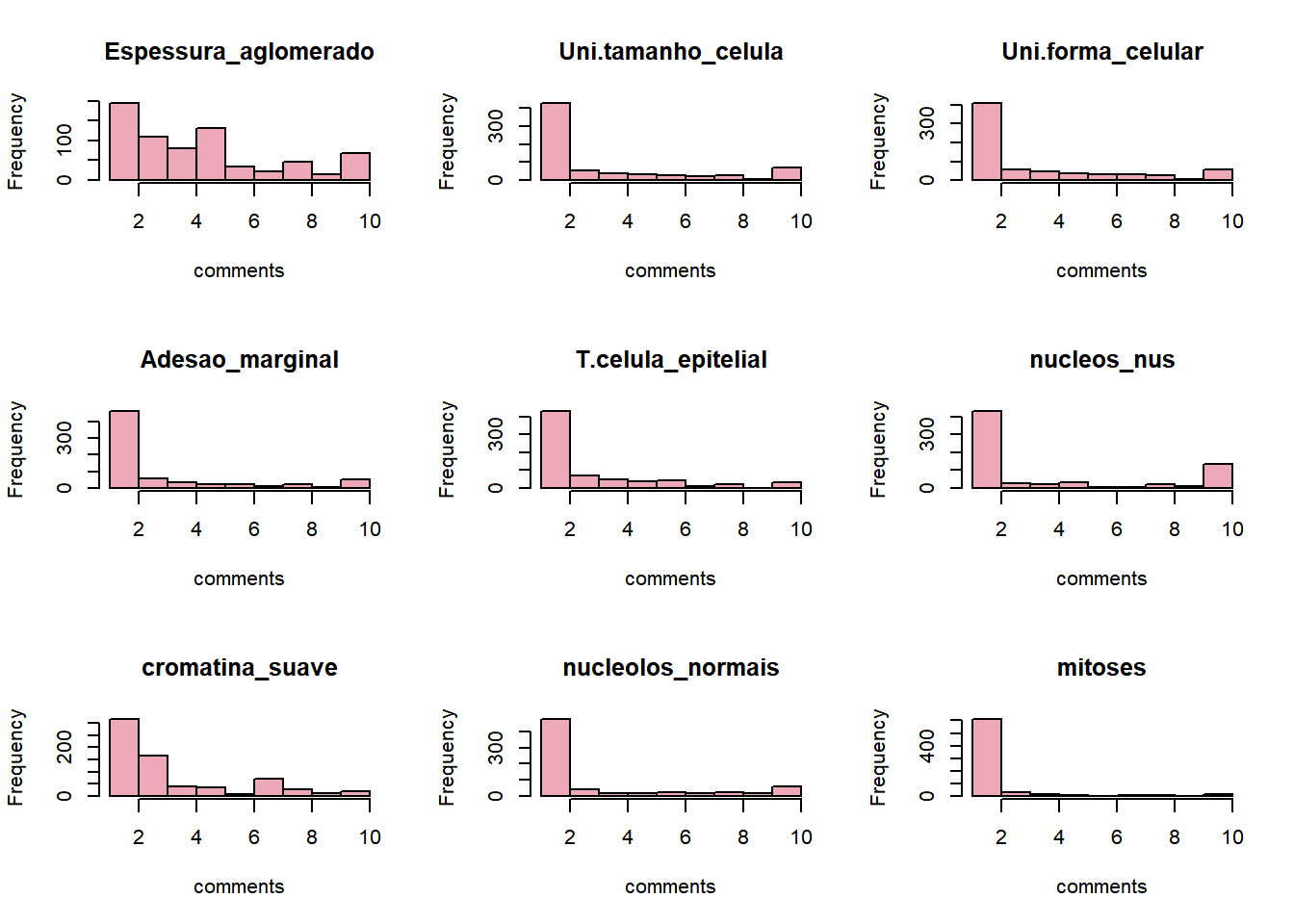
Figure 5.1: Histograma das variáveis preditoras.
Outra verificação necessária é avaliar o quanto as variáveis estão correlacionadas.
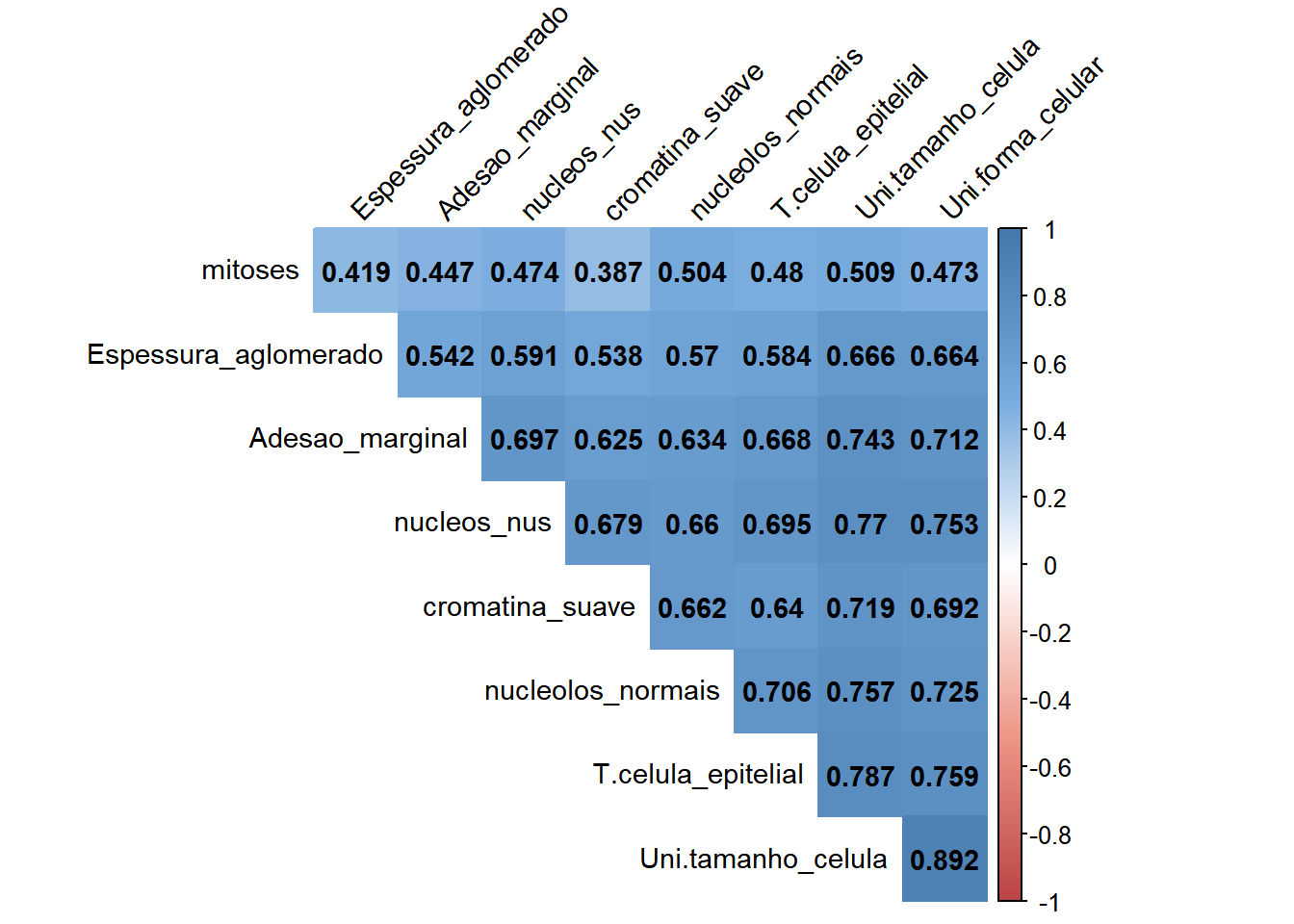
Figure 5.2: Correlação entre as variáveis preditoras.
Conforme pode ser observado na Figura 5.2 não existe nenhuma variável com correlação maior ou igual a 0.90. Outra verificação importante que deve ser feita no conjunto de dados é verificar o balanceamento desses dados em relação a variável de interesse classe.
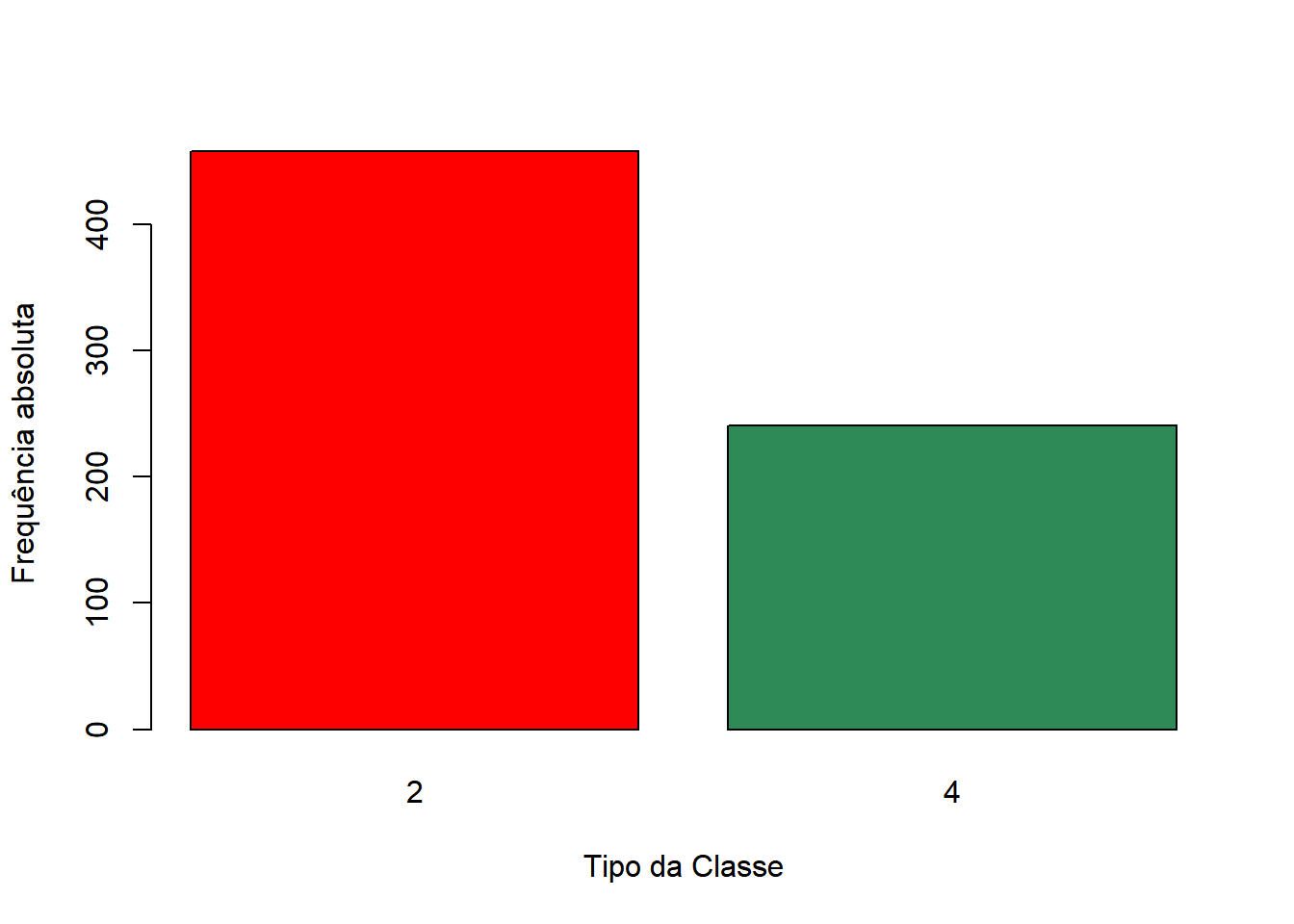
Figure 5.3: Visualização do comportamento dos dados em relação a variável Classe.
Observa-se na Figura 5.3 que a quantidade de observações da classe 2 é quase o dobro das observações da classe 4. Portanto, será necessário estratificar os dados em relação a variável classe ao realizar a divisão entre treinamento e teste.
5.3 Árvore de decisão
Decision trees (Árvores de decisão) é uma algoritmo de classificação e regressão de dados. A formação de uma árvore de decisão é composta por nós, ramos e folhas. Os nós raizes são as regiões onde se localiza os testes lógicos de separação de dados para tomada de decisão e são divididos em nó raiz (ou nó pai) e nós filhos (ou de tomada de decisão), onde geralmente é gerado uma sub-árvore. Os ramos fazem uma conexão entre os nós raiz e os nós filhos. Nas folhas se localizam os rótulos ou valores. Dessa forma o algoritmo percorre a árvore e tomada a decisão a partir dos atributos mais relevantes, na Figura 5.4 pode-se visualizar uma ilustração de como algoritmos de árvores de decisão funciona.
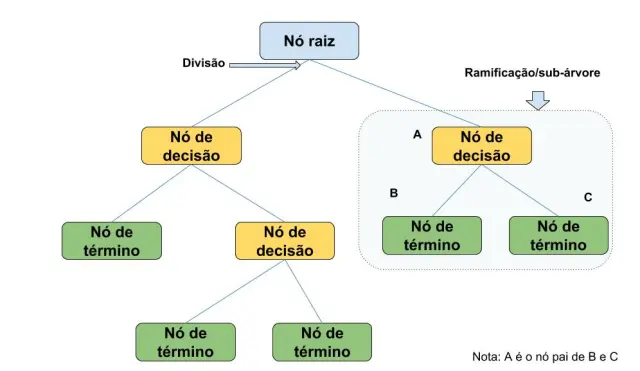
Figure 5.4: Esquema de um algoritmo de árvore de decisão.
Fonte: https://www.vooo.pro/insights/
A aplicação de qualquer método de aprendizagem de máquina, consiste em utilizar uma base de dados, sendo esta divida em treinamento e teste. O primeiro passo é treinar o algoritmo avaliar seu desempenho nos dados de treinamento, selecionar o melhor modelo segundo a métrica utilizada e em seguida testar o modelo que obtiver o melhor desempenho.
5.3.1 Divisão dos dados
Primeiro, é realizado a divisão do conjunto de dados em treino e teste. O argumento strata usa amostragem estratificada para criar a reamostragem e faz um balanceamento dos dados de modo a equilibrar a quantidade de observações do atributo classe (0 - para tumor benigno, 1 - para tumor maligno) nos conjuntos de treinamento e teste.
set.seed(1234)
divisao <- initial_split(dados, strata = classe)
divisao## <Analysis/Assess/Total>
## <523/176/699>O conjunto de dados é dividido em aproximadamente 75% dos dados para treinamento e 25% para teste, abaixo e carregado os dados para treino e teste respectivamente. Fixa-se uma semente set.seed() para que os resultados não se altere a cada execução. Sendo assim, as observações que farão parte da base de dados de treino e teste serão as mesmas, independentemente de quantas vezes o código seja executado.
data_treino <- training(divisao)
data_teste <- testing(divisao)Os dados de treinamento serão usados para ajustar o modelo e seus parâmetros, e os dados de teste serão usados para avaliar o desempenho do modelo final.
5.3.2 Pré - processamento
pre_recipe <- recipe(classe ~ ., data = data_treino) %>%
step_impute_median(all_predictors()) Primeiramente a função recipe () recebe a variável resposta classe em relação a todas as demais variáveis explicativas (.) e atribui-se os dados de treinamento. A função step_impute_median() substitui os dados faltantes do conjunto de dados pela a sua mediana. Não foi utilizado nenhum tipo de normalização/padronização dos dados pois ambas as variáveis explicativas estão em uma mesma escala de medida.
A função step_impute_knn() também existente no pacote é útil quando existe muitos dados faltantes e a alternativa e tirá-los do conjunto de dados. Neste caso optou-se pela imputação do valores faltantes pela mediana pois existia apenas 16 observações faltantes no conjunto de dados inteiro.
Observe que a receita foi salva em um objeto chamado pre_recipe que será utilizada por todos os algoritmos de classificação.
5.3.3 Validação cruzada
A validação cruzada V-fold (também conhecida como validação cruzada k-fold) divide aleatoriamente os dados em grupos V de tamanho aproximadamente iguais (chamados “dobras”). A técnica de validação cruzada usa reamostragem para comparar e selecionar os melhores modelos. O vfold_cv() já divide por padrão em 4 partes v = 4.
set.seed(1243)
val_set <- vfold_cv(data_treino, v = 4, strata = classe)5.3.4 Definição do modelo
Define-se o modelo que será utilizado, nesse caso o algoritmo de árvore de decisão, decision_tree() que recebe os argumentos cost_complexity que determina o parâmetro de custo/complexidade do modelo e min_n um número inteiro para o número mínimo de pontos de dados em um nó que são necessários para que o nó seja dividido ainda mais. Para ambos os argumentos cost_complexity e min_n atribui-se a função tune() do pacote tune que significa que os valores serão ajustados pelo pipeline, ou seja, será escolhido o modelo que apresentar o melhor resultado.
tree_model <- decision_tree(cost_complexity = tune(),
min_n = tune()) %>%
set_engine("rpart") %>%
set_mode("classification")A função set_engine() e set_mode() define o pacote que será utilizado para o ajuste, e o modelo respectivamente. O pipe (%>%) é o operador de encadeamento que é utilizado para separar e organizar funções dentro de funções, de maneira que simplificará o código.
Após a definição do modelo é criado o fluxo de trabalho workflow().
tree_workflow <- workflow() %>%
add_model(tree_model) %>%
add_recipe(pre_recipe)O modelo add_model() e o pré-processamento add_recipe() são colocados dentro de um workflow() e salvo em um objeto chamado tree_workflow.
5.3.5 Treinamento
Nessa etapa é realizado o treinamento do modelo, onde o fluxo de trabalho criado anteriormente é salvo em um objeto chamado tree_trained e é adicionado a função tune_grid() onde é adicionado a validação cruzada salva no objeto val_set.
tree_trained <- tree_workflow %>%
tune_grid(
val_set,
grid = 10,
control = control_grid(save_pred = TRUE),
metrics = metric_set(roc_auc)
)O argumento grid define a quantidade de combinações de parâmetros do modelo que será treinado, a função control_grid() armazena as informações do treinamento para serem avaliadas posteriormente. Em metric_set() define-se as métricas que serão utilizadas na avaliação dos modelos.
tree_trained %>% show_best("roc_auc")## # A tibble: 5 × 8
## cost_complexity min_n .metric .estimator mean n std_err .config
## <dbl> <int> <chr> <chr> <dbl> <int> <dbl> <chr>
## 1 0.00000000602 18 roc_auc binary 0.962 4 0.00624 Preprocessor1_Mo…
## 2 0.00000258 33 roc_auc binary 0.961 4 0.00635 Preprocessor1_Mo…
## 3 0.00000000941 39 roc_auc binary 0.960 4 0.00565 Preprocessor1_Mo…
## 4 0.000122 24 roc_auc binary 0.959 4 0.00587 Preprocessor1_Mo…
## 5 0.000000169 26 roc_auc binary 0.959 4 0.00587 Preprocessor1_Mo…Utilizando a função show_best() e definindo a métrica utilizada é possível visualizar os modelos gerados. Caso deseje visualizar todos os modelos gerados independente da métrica utlizada use collect_metrics(), conforme código abaixo.
tree_trained %>%
collect_metrics() %>%
print(n = Inf)5.3.6 Seleção do melhor modelo
Observa-se que o melhor modelo obteve uma AUC de 0.962. Usando a função select_best() define-se a métrica e é mostrado o melhor modelo.
tree_best_tune <- select_best(tree_trained, 'roc_auc')
final_tree_model <- tree_model %>%
finalize_model(tree_best_tune)
tree_best_tune## # A tibble: 1 × 3
## cost_complexity min_n .config
## <dbl> <int> <chr>
## 1 0.00000000602 18 Preprocessor1_Model10O Modelo 10 obteve o melhor resultado usando a métrica de Curva Roc, escolhe-se então esse modelo como modelo final e aplica-se os dados de teste ao modelo.
5.3.7 Aplicando os dados de teste ao modelo
Na etapa final é criado o novo fluxo de trabalho e é submetido os dados de teste ao modelo escolhido a partir dos dados de treinamento. É adicionado em add_recipe() o pré-processamento dos dados criado no início, em add_model() é adicionado o modelo final. A função last_fit() indica que é último ajuste a ser realizado.
previsoes <- workflow() %>%
add_recipe(pre_recipe) %>%
add_model(final_tree_model) %>%
last_fit(divisao)Usando collect_predictions() visualizamos as predições salva no objeto previsoes.
previsoes %>% collect_predictions()## # A tibble: 176 × 7
## id .pred_2 .pred_4 .row .pred_class classe .config
## <chr> <dbl> <dbl> <int> <fct> <fct> <chr>
## 1 train/test split 0.979 0.0210 1 2 2 Preprocessor1_Mode…
## 2 train/test split 0.979 0.0210 3 2 2 Preprocessor1_Mode…
## 3 train/test split 0.979 0.0210 5 2 2 Preprocessor1_Mode…
## 4 train/test split 0.979 0.0210 8 2 2 Preprocessor1_Mode…
## 5 train/test split 0.979 0.0210 9 2 2 Preprocessor1_Mode…
## 6 train/test split 0.979 0.0210 10 2 2 Preprocessor1_Mode…
## 7 train/test split 0.979 0.0210 12 2 2 Preprocessor1_Mode…
## 8 train/test split 0.0556 0.944 19 4 4 Preprocessor1_Mode…
## 9 train/test split 0.105 0.895 21 4 4 Preprocessor1_Mode…
## 10 train/test split 0.105 0.895 26 4 4 Preprocessor1_Mode…
## # … with 166 more rowsEsse resultado podem ser visualizado atráves de uma matriz de confusão (Figura 5.5), onde é possível avaliar a quantidade de previsões certas e erradas que o modelo obteve.
previsoes %>% collect_predictions() %>%
conf_mat(truth = classe,
estimate = .pred_class, dnn = c("Previsto", "Real")) %>%
autoplot(type = "heatmap") +
scale_fill_gradient(low = "pink3", high = "cyan4")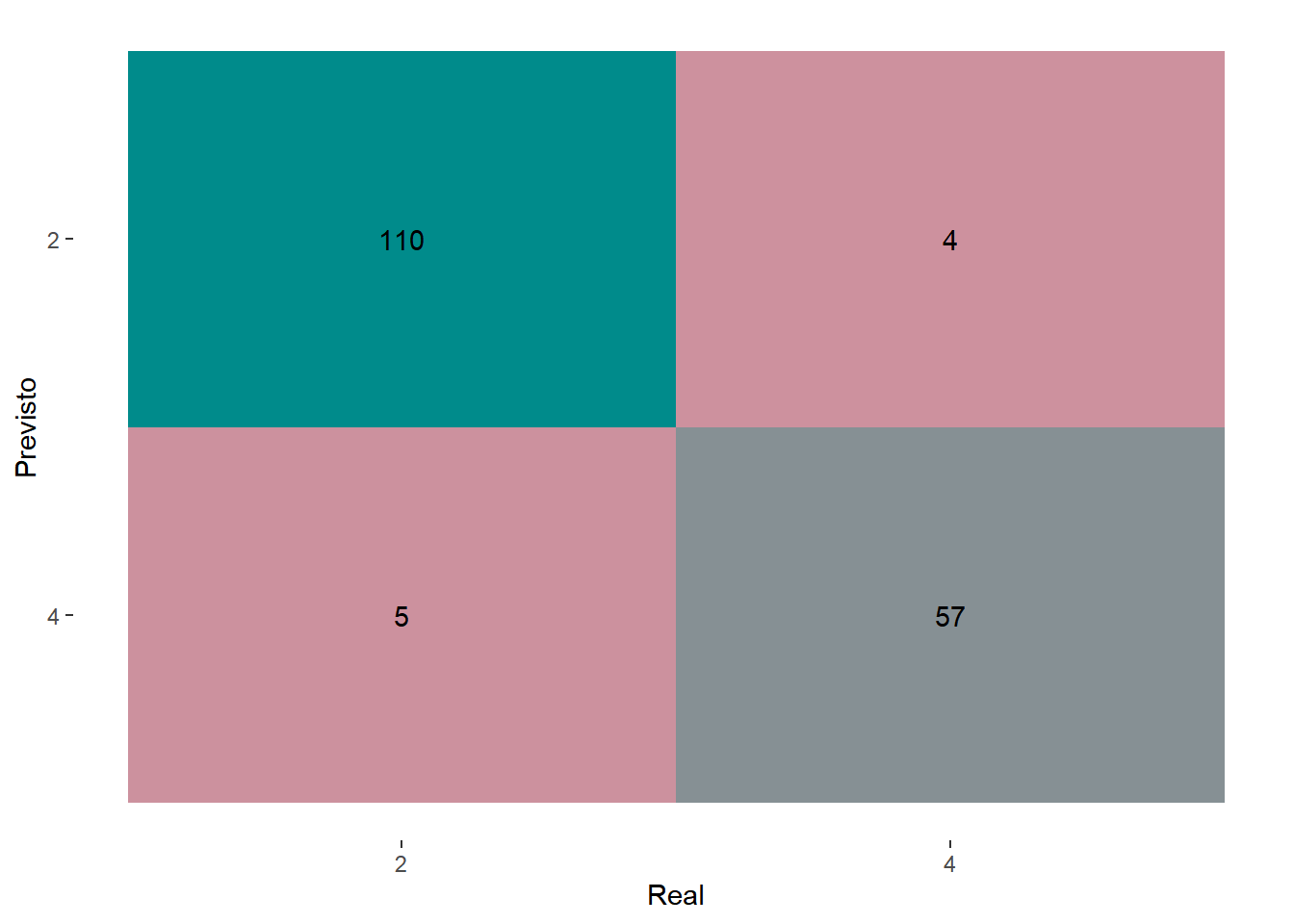
Figure 5.5: Matriz de confusão para o algoritmo de árvore de decisão.
Pode-se também visualizar o resultado final atráves de uma Curva Roc (Figura 5.6) usando a função roc_curve() e pedindo o autoplot().
previsoes %>% collect_predictions() %>%
roc_curve(classe, .pred_2) %>%
autoplot()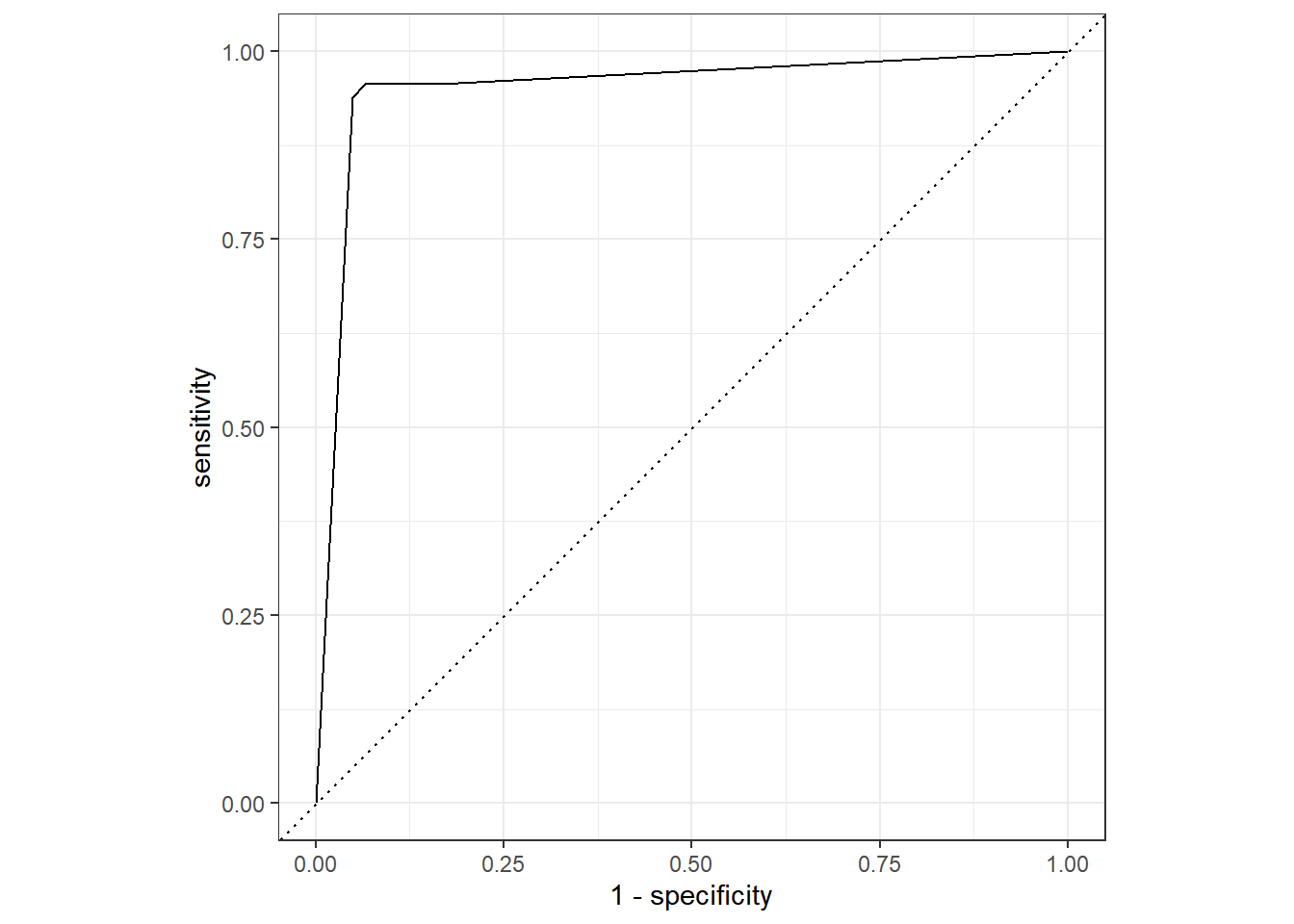
Figure 5.6: Desempenho do algoritmo Árvore de decisão nos dados de teste.
Quanto maior for a distância entre a curva e a linha, melhor é o desempenho do algoritmo. Como podemos visualizar o desempenho do modelo foi satisfatório. É possível obter o valor da AUC para o dados de teste usando novamente a função show_best() nos dados de teste.
previsoes %>% show_best("roc_auc") ## # A tibble: 1 × 7
## .workflow .metric .estimator mean n std_err .config
## <list> <chr> <chr> <dbl> <int> <dbl> <chr>
## 1 <workflow> roc_auc binary 0.951 1 NA Preprocessor1_Model1Obtendo a AUC do modelo nos dados de teste de outra maneira.
previsoes %>% collect_predictions() %>%
roc_auc(classe, .pred_2)## # A tibble: 1 × 3
## .metric .estimator .estimate
## <chr> <chr> <dbl>
## 1 roc_auc binary 0.951Comparando o valor da AUC tanto nos dados de treinamento como de teste, observa-se um desempenho similar.
O processo de aplicação do algoritmo de Machine Learning para o demais modelos seguem a mesma lógica do que foi apresentado para o modelo de árvore de decisão, a única mudança que ocorre é na definição do modelo.
5.4 Random Forest
O algoritmo Random Forest é formado por várias árvores de decisão, geralmente treinados com o método de bagging, que gera um conjunto de dados por amostragem bootstrap, a ideia principal do método é criar combinações de modelos que aumentem o resultado final.
O modelo de Random Forest tanto pode ser utilizado para classificação como para regressão. Um dos principais benefícios do algoritmo é a possibilidade de se trabalhar com um grande conjunto de dados de maior dimensão.
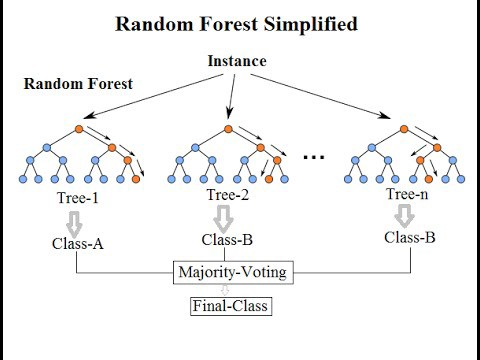
Figure 5.7: Exemplo do funcionamento do algoritmo Random Forest.
Fonte: https://estatsite.com.br/
Diferentemente da árvore de decisão, o funcionamento do Random Forest consiste no crescimento de várias árvores, onde cada árvore fornece uma classificação de votos para a classe, até chegar ao resultado final, conforme é mostrado na Figura 5.7.
Algoritmos baseaddos em árvore de decisão utilizam a média em regressão e os votos da maioria na classificação para dar uma resposta final.
5.4.1 Definição do modelo
Define-se o modelo que será utilizado, nesse caso é o algoritmo Random Forest rand_forest() que recebe os argumentos mtry que determina o número de preditores que serão amostrados ateatoriamente em cada divisão, trees que determina o número de árvores contidas no conjunto e min_n um número inteiro para o número mínimo de pontos de dados em um nó que são necessários para que o nó seja dividido ainda mais. Para os argumentos mtry e min_n atribui-se a função tune() que significa que os valores serão ajustados pelo pipeline, para trees foi atribuido o total de 1000 árvores.
library(ranger)
rf_model <- rand_forest(mtry = tune(), min_n = tune(),
trees = 1000) %>%
set_engine("ranger") %>%
set_mode("classification")Após a definição do modelo é criado o fluxo de trabalho workflow(), em add_recipe() adciona o pre_recipe que é a receita criada na parte de pré-processamento feito antes da aplicação do modelo de árvore de decisão mostrado na seção 5.3.
rf_workflow <- workflow() %>%
add_model(rf_model) %>%
add_recipe(pre_recipe)5.4.2 Treinamento
Nessa etapa é realizado o treinamento do modelo para o algoritmo Random Forest.
rf_trained <- rf_workflow %>%
tune_grid(
val_set,
grid = 10,
control = control_grid(save_pred = TRUE),
metrics = metric_set(roc_auc)
)Observa-se os modelos gerados a partir do algoritmo de Random Forest.
rf_trained %>% show_best("roc_auc")## # A tibble: 5 × 8
## mtry min_n .metric .estimator mean n std_err .config
## <int> <int> <chr> <chr> <dbl> <int> <dbl> <chr>
## 1 4 19 roc_auc binary 0.994 4 0.00207 Preprocessor1_Model01
## 2 2 28 roc_auc binary 0.994 4 0.00214 Preprocessor1_Model09
## 3 5 9 roc_auc binary 0.994 4 0.00199 Preprocessor1_Model07
## 4 1 32 roc_auc binary 0.993 4 0.00250 Preprocessor1_Model04
## 5 6 14 roc_auc binary 0.993 4 0.00216 Preprocessor1_Model065.4.3 Seleção do melhor modelo
Usando a função select_best() e defininado a métrica é mostrado o melhor modelo, dentre todos os modelos gerados.
rf_best_tune <- select_best(rf_trained, 'roc_auc')
final_rf_model <- rf_model %>%
finalize_model(rf_best_tune)
rf_best_tune## # A tibble: 1 × 3
## mtry min_n .config
## <int> <int> <chr>
## 1 4 19 Preprocessor1_Model01O modelo 1 aprensentou o melhor resultado com uma AUC de 0.994.
5.4.4 Aplicando os dados de teste ao modelo
O código abaixo retorna as predições obtidas para os dados de teste e é salvo em um objeto chamado previsoes.
previsoes <- workflow() %>%
add_recipe(pre_recipe) %>%
add_model(final_rf_model) %>%
last_fit(divisao) Para visualizar as previsões utiliza-se a função collect_predictions(), onde
em classe é mostrado os valores reais e em .pred_class os valores preditos.
previsoes %>% collect_predictions()## # A tibble: 176 × 7
## id .pred_2 .pred_4 .row .pred_class classe .config
## <chr> <dbl> <dbl> <int> <fct> <fct> <chr>
## 1 train/test split 1 0 1 2 2 Preprocessor1_Mod…
## 2 train/test split 1.00 0.000119 3 2 2 Preprocessor1_Mod…
## 3 train/test split 0.997 0.00329 5 2 2 Preprocessor1_Mod…
## 4 train/test split 1 0 8 2 2 Preprocessor1_Mod…
## 5 train/test split 0.829 0.171 9 2 2 Preprocessor1_Mod…
## 6 train/test split 1 0 10 2 2 Preprocessor1_Mod…
## 7 train/test split 1 0 12 2 2 Preprocessor1_Mod…
## 8 train/test split 0.00307 0.997 19 4 4 Preprocessor1_Mod…
## 9 train/test split 0.361 0.639 21 4 4 Preprocessor1_Mod…
## 10 train/test split 0.354 0.646 26 4 4 Preprocessor1_Mod…
## # … with 166 more rowsVisualizado a matriz de confusão para o modelo escolhido (Figura 5.8).
previsoes %>% collect_predictions() %>%
conf_mat(truth = classe,
estimate = .pred_class, dnn = c("Previsto", "Real")) %>%
autoplot(type = "heatmap") +
scale_fill_gradient(low = "pink3", high = "cyan4")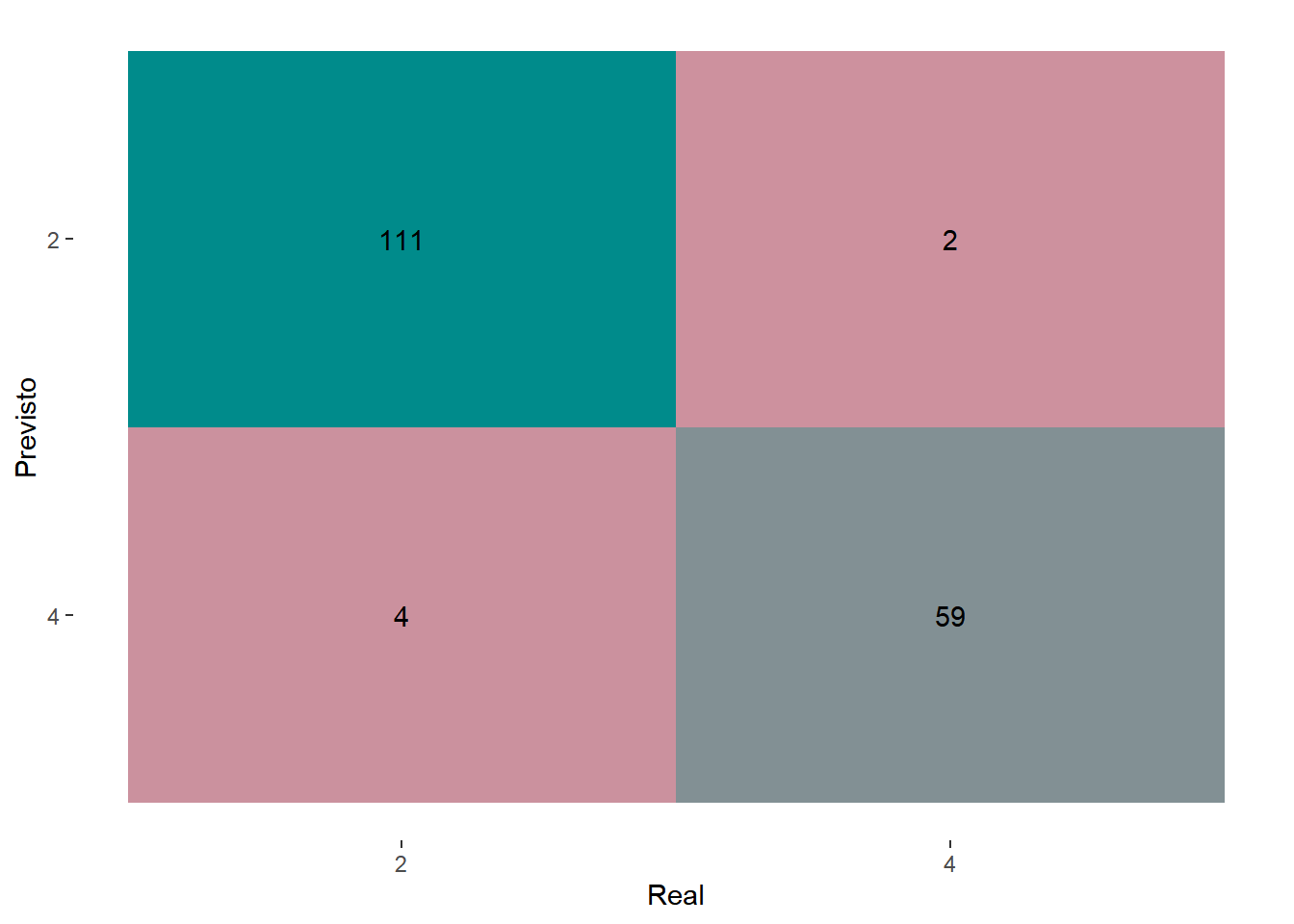
Figure 5.8: Matriz de confusão para o algoritmo de Random Forest.
A seguir tem-se o código que gera a curva Roc, conforme pode ser observado na Figura 5.9, onde é mostrado o desempenho do modelo nos dados de teste.
previsoes %>% collect_predictions() %>%
roc_curve(classe, .pred_2) %>%
autoplot()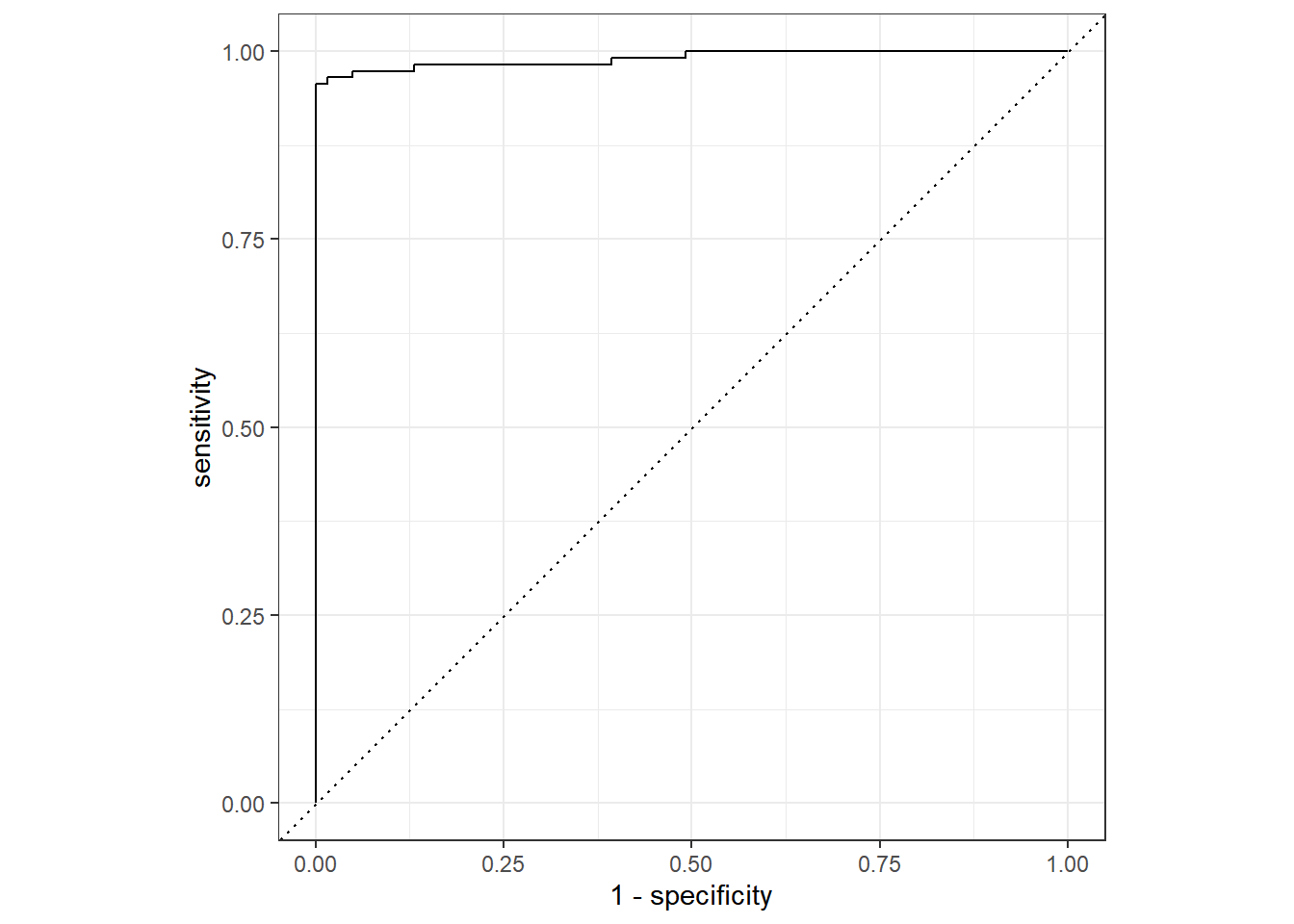
Figure 5.9: Desempenho do algoritmo Random Forest nos dados de teste.
Obtendo a AUC da Curva ROC.
previsoes %>% collect_predictions() %>%
roc_auc(classe, .pred_2)## # A tibble: 1 × 3
## .metric .estimator .estimate
## <chr> <chr> <dbl>
## 1 roc_auc binary 0.991Observe que o algoritmo Random Forest obteve melhores resultados se comparado com algoritmo de árvore de decisão.
5.5 Aprendizagem baseada em instâncias (KNN)
O K-Nearest Neighbors (KNN) ou simplesmente K vizinhos mais próximos, traduzindo para o português, é um algoritmo que como o próprio nome diz, classifica os novos dados com base em uma medida de similaridade entre seus “vizinhos” mais próximos, ou seja, forma grupos com os atributos que possuem características semelhantes. O KNN faz a comparação das distâncias entre os dados no espaço euclidiano.
A aprendizagem baseada em instâncias é conhecida como sendo uma aprendizagem preguiçosa, a lentidão ocorre à medida que o volume de dados aumenta, tornando a sua escolha impraticável em situações em que as previsões precisam ser feitas rapidamente. É uma algoritmo majoriatariamente utilizado para classificação.
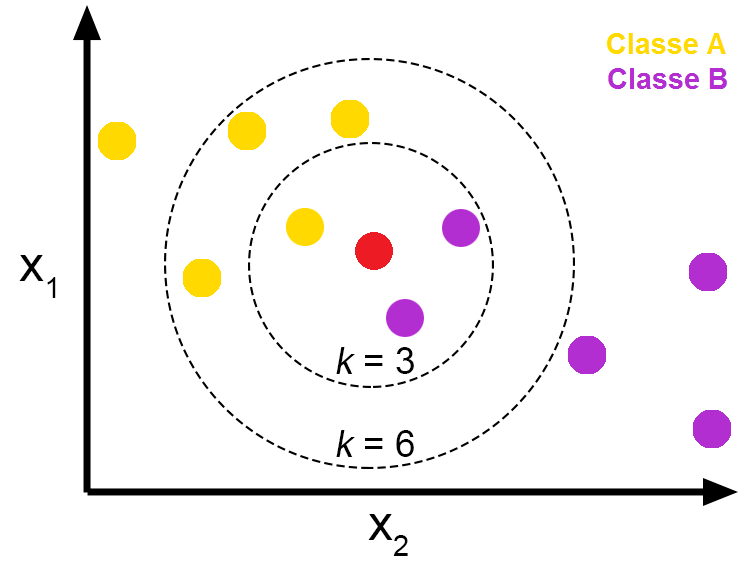
Figure 5.10: Exemplo do funcionamento do algoritmo KNN.
Fonte: https://medium.com/
Conforme mostra a Figura 5.10, o algoritmo KNN pressupõe que itens semelhantes estão próximos um dos outros, então tenta encaixar o novo dado nos conjuntos de seus vizinhos mais próximos em relação a similaridade existe entre eles. O cálculo de distância mais comum é a distância euclidiana, mas outras distâncias podem ser utilizadas.
5.5.1 Definição do modelo
Define-se o modelo que será utilizado, nesse caso é o algoritmo KNN nearest_neighbor() que recebe os argumentos neighbors que determina o número de vizinhos a serem considerados, weight_func usado para ponderar as distâncias entre as amostras. Para ambos os parâmetros atribui-se a função tune() que significa que os valores serão ajustados pelo pipeline.
library(kknn)
knn_model <- nearest_neighbor(neighbors = tune(),
weight_func = tune()) %>%
set_engine("kknn") %>%
set_mode("classification")Após a definição do modelo é criado o fluxo de trabalho workflow().
knn_workflow <- workflow() %>%
add_model(knn_model) %>%
add_recipe(pre_recipe)O modelo add_model() e o pré-processamento add_recipe() são colocados dentro de um workflow() e salvo em um objeto chamado knn_workflow.
5.5.2 Treinamento
Nessa etapa é realizado o treinamento do modelo para o algoritmo KNN.
knn_trained <- knn_workflow %>%
tune_grid(
val_set,
grid = 10,
control = control_grid(save_pred = TRUE),
metrics = metric_set(roc_auc)
)Visualização dos resultados usando para métrica de Curva ROC.
knn_trained %>% show_best("roc_auc")## # A tibble: 5 × 8
## neighbors weight_func .metric .estimator mean n std_err .config
## <int> <chr> <chr> <chr> <dbl> <int> <dbl> <chr>
## 1 9 inv roc_auc binary 0.988 4 0.00632 Preprocessor1_M…
## 2 10 gaussian roc_auc binary 0.988 4 0.00646 Preprocessor1_M…
## 3 14 epanechnikov roc_auc binary 0.987 4 0.00623 Preprocessor1_M…
## 4 12 cos roc_auc binary 0.987 4 0.00611 Preprocessor1_M…
## 5 13 triweight roc_auc binary 0.986 4 0.00565 Preprocessor1_M…Observa-se o modelos gerados conforme definido na etapa de treinamento.
5.5.3 Seleção do melhor modelo
knn_best_tune <- select_best(knn_trained, 'roc_auc')
final_knn_model <- knn_model %>%
finalize_model(knn_best_tune)
knn_best_tune## # A tibble: 1 × 3
## neighbors weight_func .config
## <int> <chr> <chr>
## 1 9 inv Preprocessor1_Model05O modelo 5 apresentou melhores resultados com AUC de 0.988, utilizando o algortimo KNN.
5.5.4 Aplicando os dados de teste ao modelo
previsoes <- workflow() %>%
add_recipe(pre_recipe) %>%
add_model(final_knn_model) %>%
last_fit(divisao) previsoes %>% collect_predictions()## # A tibble: 176 × 7
## id .pred_2 .pred_4 .row .pred_class classe .config
## <chr> <dbl> <dbl> <int> <fct> <fct> <chr>
## 1 train/test split 1 0 1 2 2 Preprocessor1_Mode…
## 2 train/test split 1 0 3 2 2 Preprocessor1_Mode…
## 3 train/test split 1 0 5 2 2 Preprocessor1_Mode…
## 4 train/test split 1 0 8 2 2 Preprocessor1_Mode…
## 5 train/test split 0.882 0.118 9 2 2 Preprocessor1_Mode…
## 6 train/test split 1 0 10 2 2 Preprocessor1_Mode…
## 7 train/test split 1 0 12 2 2 Preprocessor1_Mode…
## 8 train/test split 0 1 19 4 4 Preprocessor1_Mode…
## 9 train/test split 0.104 0.896 21 4 4 Preprocessor1_Mode…
## 10 train/test split 0.422 0.578 26 4 4 Preprocessor1_Mode…
## # … with 166 more rowsObserva-se as classificações reais dos dados de teste em classe e os valores preditos em .pred_class. Visualiza-se na Figura 5.11 a matriz de confusão para o modelo.
previsoes %>% collect_predictions() %>%
conf_mat(truth = classe,
estimate = .pred_class, dnn = c("Previsto", "Real")) %>%
autoplot(type = "heatmap") +
scale_fill_gradient(low = "pink3", high = "cyan4")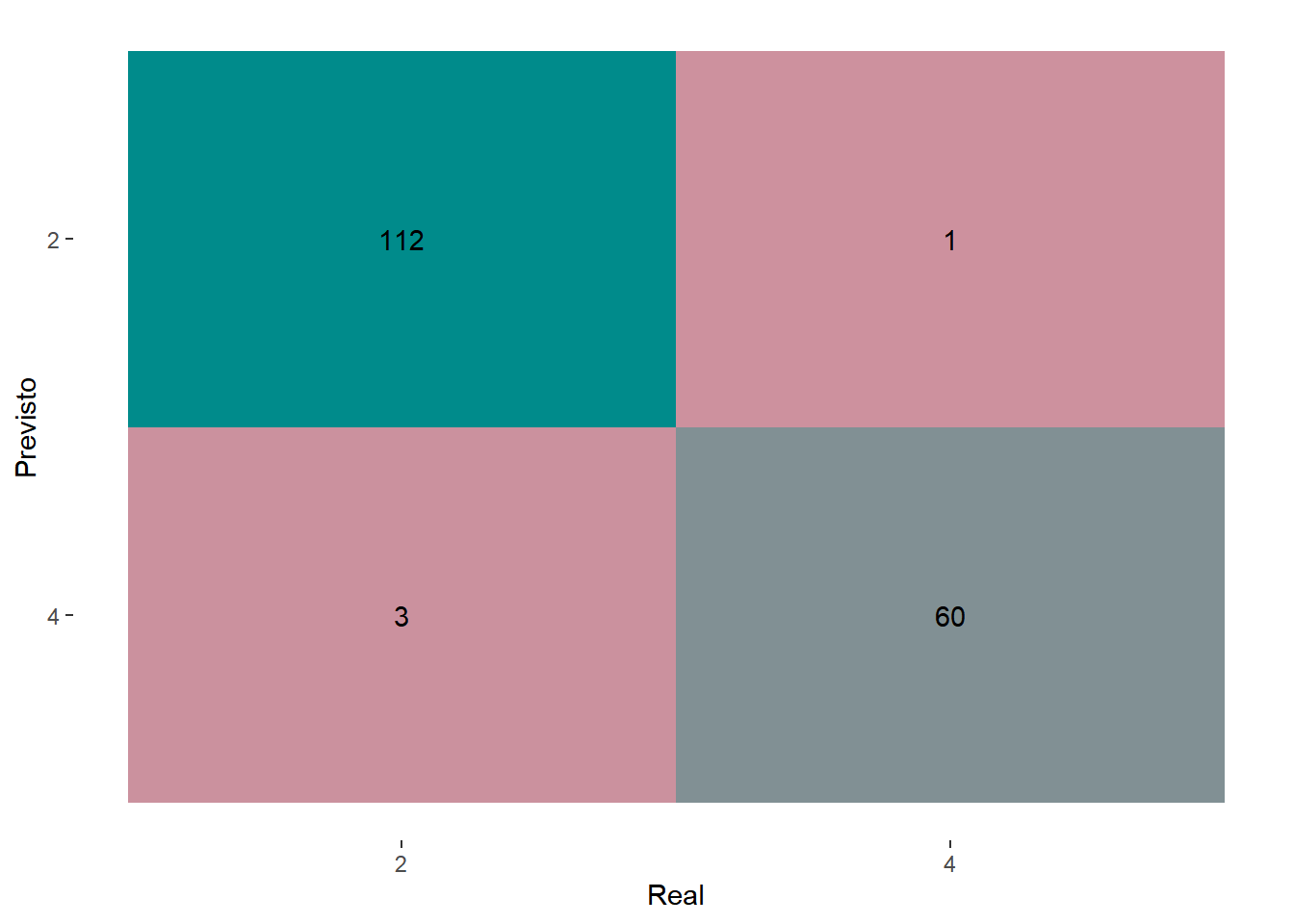
Figure 5.11: Matriz de confusão para o algoritmo KNN.
Visualizando a Curva ROC do modelo aplicado aos dados de teste (Figura 5.12).
previsoes %>% collect_predictions() %>%
roc_curve(classe, .pred_2) %>%
autoplot()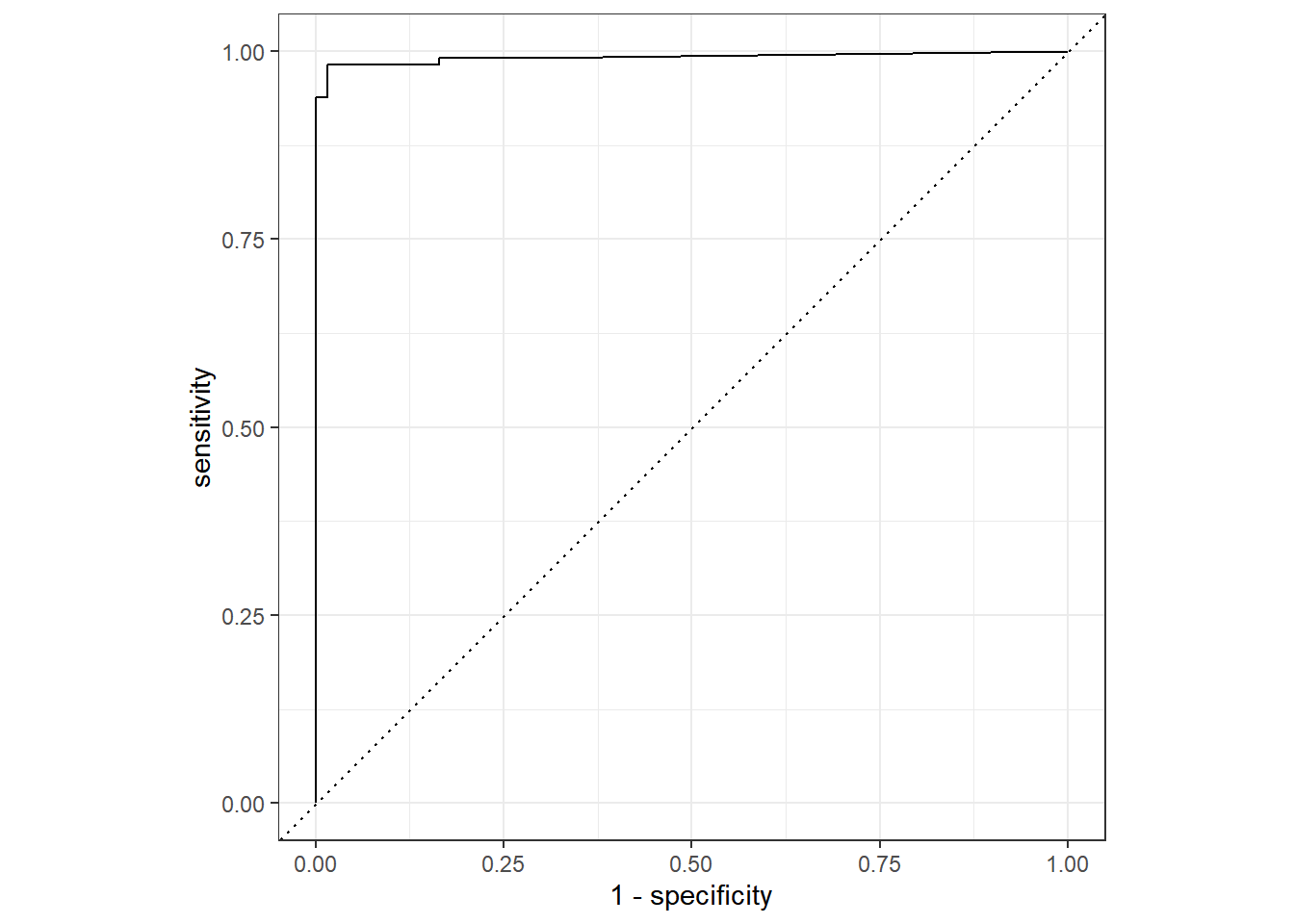
Figure 5.12: Desempenho do algoritmo KNN nos dados de teste.
Obtendo a AUC da Curva ROC para o modelo escolhido.
previsoes %>% collect_predictions() %>%
roc_auc(classe, .pred_2)## # A tibble: 1 × 3
## .metric .estimator .estimate
## <chr> <chr> <dbl>
## 1 roc_auc binary 0.992Observa-se que os resultados obtidos quando aplicado o modelo gerado pelo algoritmo KNN ao dados de teste, são satisfatórios. O algoritmo KNN teve um desempenho ainda melhor que o algoritmo de árvore de decisão.
5.6 Regressão Logística
A Regressão Logística lida apenas com problemas de classificação, apesar do nome regressão, a Regressão Logística não é um modelo que prevê valores como a Regressão Linear. A utilização da regressão logística é comum na classificação de duas classes, mas ela não se limita apenas a isso.
A Regressão Logística trabalha com conceitos de estatística e probabilidade e seu uso é comum em algoritmos de Machine learning devido criar previsões precisas no que se refere a classificação. A variável resposta deve ser dicotômica ou binária.
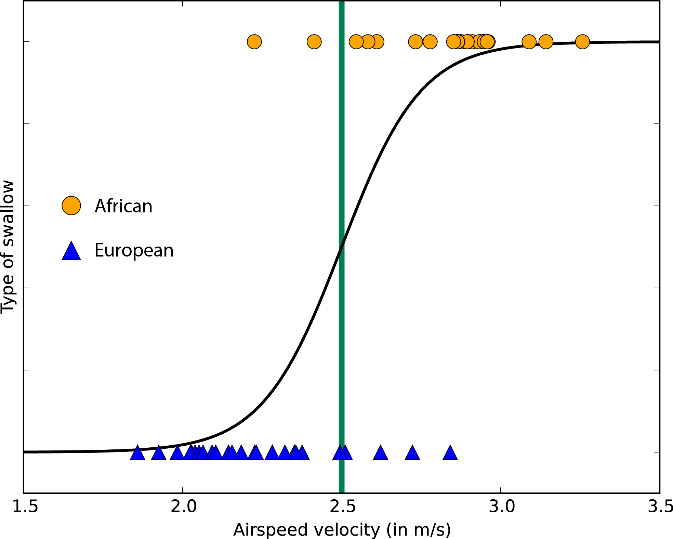
Figure 5.13: Exemplo do funcionamento da Regressão Logística.
Fonte: https://pcodinomebzero.neocities.org/
Conforme mostra a Figura 5.13, diferentemente da regressão linear o gráfico da regressão logística mostra um padrão em S e apresenta valores no intervalo de 0 a 1, ao invés de uma reta linear.
5.6.1 Definição do modelo
Define-se o modelo que será utilizado, nesse caso é o modelo de Regressão Logística logistic_reg() que recebe os argumentos penalty que é o número não negativo que representa a quantidade total de regularização e mixture um número entre zero e um que é a proporção de regularização no modelo. Para ambos os parâmetros atribui-se a função tune() que significa que os valores serão ajustados pelo pipeline.
library(glmnet)
Log_model <- logistic_reg(penalty = tune(),
mixture = tune()) %>%
set_engine("glmnet")A função set_engine()define o pacote que será utilizado para o ajuste, por se tratar de um modelo apenas de classificação não é necessário definir o tipo do modelo.
Após a definição do modelo é criado o fluxo de trabalho workflow().
Log_workflow <- workflow() %>%
add_model(Log_model) %>%
add_recipe(pre_recipe)O modelo add_model() e o pré-procesamento add_recipe() são colocados dentro de um workflow() e salvo em um objeto chamado Log_workflow.
5.6.2 Treinamento
Nessa etapa é realizado o treinamento do modelo para o algoritmo de Regressão Logística.
Log_trained <- Log_workflow %>%
tune_grid(
val_set,
grid = 10,
control = control_grid(save_pred = TRUE),
metrics = metric_set(roc_auc)
)Em todos os métodos de aprendizagem utilizaremos a mesma métrica. Usando a função show_best visualiza-se os modelos gerados.
Log_trained %>% show_best("roc_auc")## # A tibble: 5 × 8
## penalty mixture .metric .estimator mean n std_err .config
## <dbl> <dbl> <chr> <chr> <dbl> <int> <dbl> <chr>
## 1 1.02e- 1 0.804 roc_auc binary 0.995 4 0.00130 Preprocessor1_Model08
## 2 1.11e- 2 0.629 roc_auc binary 0.995 4 0.00196 Preprocessor1_Model07
## 3 3.01e- 3 0.985 roc_auc binary 0.995 4 0.00217 Preprocessor1_Model10
## 4 6.04e-10 0.0718 roc_auc binary 0.994 4 0.00197 Preprocessor1_Model01
## 5 3.10e- 4 0.333 roc_auc binary 0.994 4 0.00210 Preprocessor1_Model035.6.3 Seleção do melhor modelo
Log_best_tune <- select_best(Log_trained, 'roc_auc')
final_Log_model <- Log_model %>%
finalize_model(Log_best_tune)
Log_best_tune## # A tibble: 1 × 3
## penalty mixture .config
## <dbl> <dbl> <chr>
## 1 0.102 0.804 Preprocessor1_Model08O Modelo 8 apresentou melhores resultados com AUC de 0.995 utilizando o algoritmo de Regressão Logística.
5.6.4 Aplicando os dados de teste ao modelo
Aplicação do modelo nos dados de teste, onde os resultados são salvos em um objeto chamado previsoes.
previsoes <- workflow() %>%
add_recipe(pre_recipe) %>%
add_model(final_Log_model) %>%
last_fit(divisao) Obtendo as previsões.
previsoes %>% collect_predictions()## # A tibble: 176 × 7
## id .pred_2 .pred_4 .row .pred_class classe .config
## <chr> <dbl> <dbl> <int> <fct> <fct> <chr>
## 1 train/test split 0.878 0.122 1 2 2 Preprocessor1_Mode…
## 2 train/test split 0.881 0.119 3 2 2 Preprocessor1_Mode…
## 3 train/test split 0.883 0.117 5 2 2 Preprocessor1_Mode…
## 4 train/test split 0.896 0.104 8 2 2 Preprocessor1_Mode…
## 5 train/test split 0.921 0.0791 9 2 2 Preprocessor1_Mode…
## 6 train/test split 0.883 0.117 10 2 2 Preprocessor1_Mode…
## 7 train/test split 0.914 0.0857 12 2 2 Preprocessor1_Mode…
## 8 train/test split 0.130 0.870 19 4 4 Preprocessor1_Mode…
## 9 train/test split 0.312 0.688 21 4 4 Preprocessor1_Mode…
## 10 train/test split 0.543 0.457 26 2 4 Preprocessor1_Mode…
## # … with 166 more rowsObserva-se as classificações reais dos dados de teste em classe e os valores preditos em .pred_class. A Figura 5.14 mostra o resultado na matriz de confusão para o modelo.
previsoes %>% collect_predictions() %>%
conf_mat(truth = classe,
estimate = .pred_class, dnn = c("Previsto", "Real")) %>%
autoplot(type = "heatmap") +
scale_fill_gradient(low = "pink3", high = "cyan4")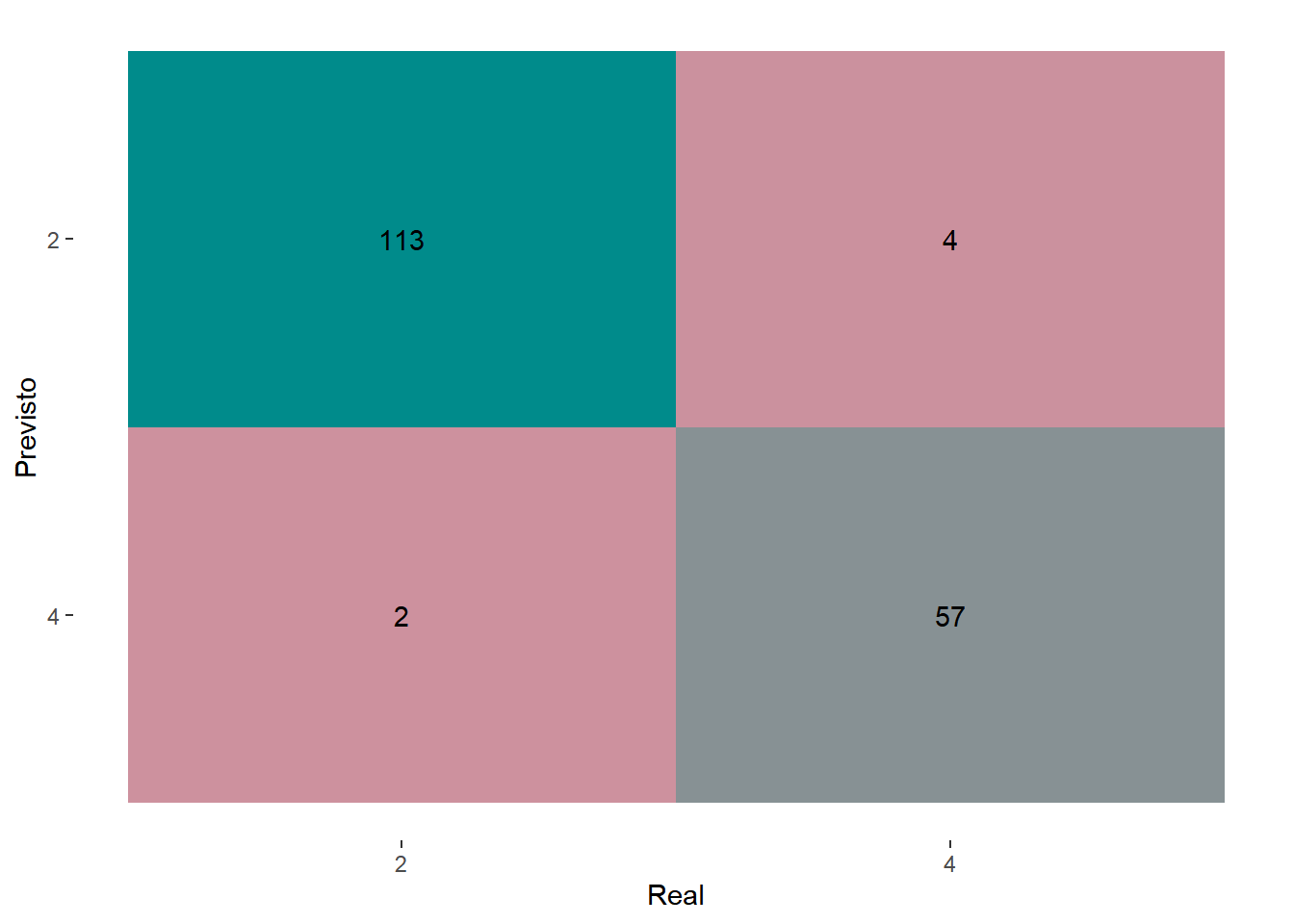
Figure 5.14: Matriz de confusão para o algoritmo de Regressão Logística.
A Figura 5.15 mostra a Curva ROC para o modelo de Regressão Logística.
previsoes %>% collect_predictions() %>%
roc_curve(classe, .pred_2) %>%
autoplot()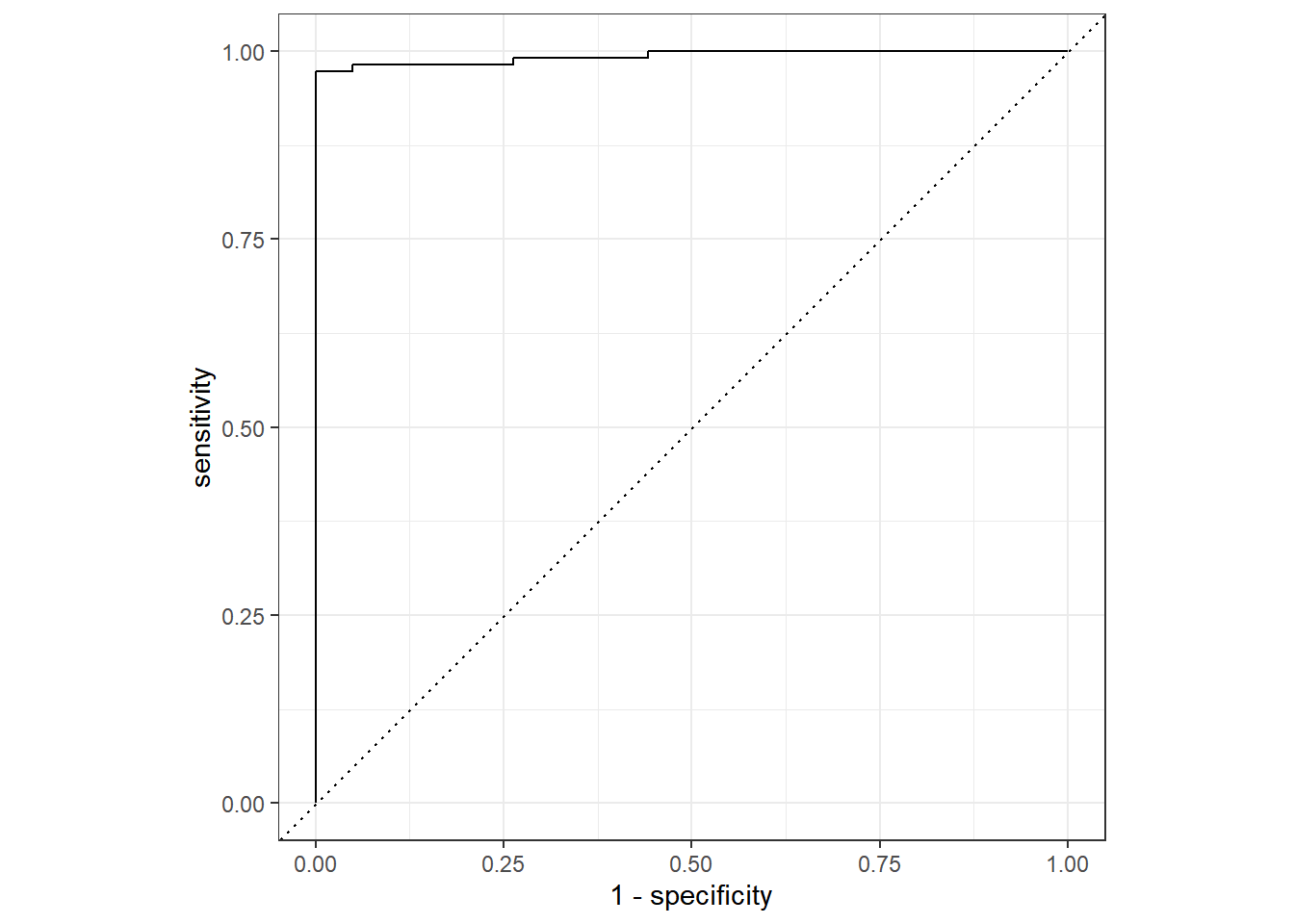
Figure 5.15: Desempenho do algoritmo Regressão Logística nos dados de teste.
Obtendo a AUC da curva ROC para o modelo selecionado.
previsoes %>% collect_predictions() %>%
roc_auc(classe, .pred_2)## # A tibble: 1 × 3
## .metric .estimator .estimate
## <chr> <chr> <dbl>
## 1 roc_auc binary 0.993O modelo de Regressão Logística teve um resultado semelhante ao algoritmo Random Forest, porém com um desempenho um pouco melhor considerando o valor da AUC. A Tabela 5.1 fornece um resumo dos resultados.
| Algoritmos | Modelo | AUC_treino | Erro | AUC_teste |
|---|---|---|---|---|
| Árvore de decisão | Modelo 10 | 0.962 | 0.00624 | 0.951 |
| Random Forest | Modelo 1 | 0.994 | 0.00207 | 0.991 |
| Algoritmo KNN | Modelo 5 | 0.988 | 0.00632 | 0.992 |
| Regressão Logística | Modelo 8 | 0.995 | 0.00130 | 0.993 |
Comparando os resultados dos modelos utilizados, aquele que teve um melhor desempenho com AUC de 0.995 foi algoritmo de Regressão Logística se comparado com os demais modelos. O modelo de Regressão Logística também foi o que generalizou melhor quando aplicado nos dados de teste.