第 27 章 ggplot2之统计图层
27.1 导言
美学映射是图形语法中非常重要的一个概念,变量映射到视觉元素,然后通过几何形状GEOM画出图形。(下图是每个几何形状所对应的视觉元素)
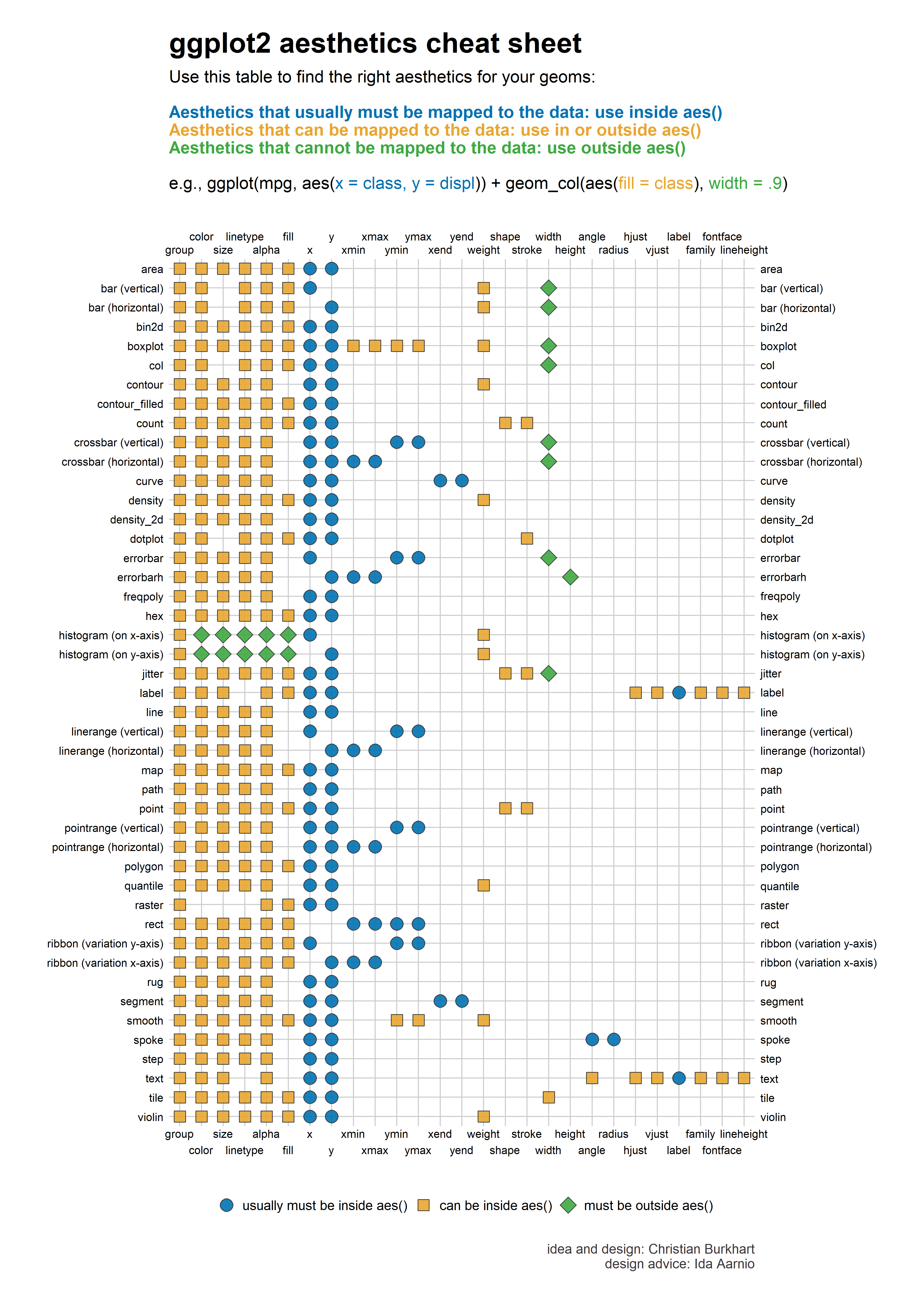
图 27.1: ggplot2中的几何形状与美学映射
比如geom_point(mapping = aes(x = mass, y = height)) 将会画出散点图,这里的x轴代表mass变量,而y轴代表height变量.
因为geom_*()很强大而且也很容易理解,所以一般我们不会去思考我们的数据在喂给ggplot()后发生了什么,只希望能出图就行了。比如下面的直方图例子
library(tidyverse)
library(palmerpenguins)
ggplot(data = penguins, mapping = aes(x = body_mass_g)) +
geom_histogram()
这里发生了什么呢?你可能看到body_mass_g这个变量代表了x轴,这个没错,但想弄清楚这个直方图,需要回答下面的问题
- 映射到
x轴的变量被分成了若干离散的小区间(bins) - 需要计算每个小区间中有多少观测值落入其中
- 用于
y轴上是一个新的变量 - 最终,用户提供的
x变量和经过计算处理后的y变量,共同确定了柱状图中每个柱子的位置和高度
我并不是说,不能给出geom_histogram()详细说明就是一个傻子。相反,我这里的本意是强调数据->视觉元素的映射并不是理所当然的,尽管看上去往往非常自然、直观和客观。
我们这里是提醒下,我们是否想过,修改上面中间过程,比如第1步和第2步,然后看看输出的图形是否还是直方图。
这个想法非常重要,但我们很少想到。某种程度是因为在我们最初学习ggplot画图的时候,ggplot已经影响了我们的思维方式。比如,初学者可能经历过拿到数据却还不出图形的受挫感,举个例子来说,这里有个数据
d <- tibble::tribble(
~variable, ~subject1, ~subject2, ~subject3,
"mass", 75, 70, 55,
"height", 154, 172, 144
)
d## # A tibble: 2 × 4
## variable subject1 subject2 subject3
## <chr> <dbl> <dbl> <dbl>
## 1 mass 75 70 55
## 2 height 154 172 144用geom_point(aes(x = mass, y = height)) 画图,却报错了。初学者可能苦苦搜索答案,然后被告知,ggplot画图需要先弄成tidy格式
d %>% pivot_longer(
cols = subject1:subject3,
names_to = "subject",
names_pattern = "subject(\\d)",
values_to = "value"
) %>%
pivot_wider(names_from = variable,
values_from = value)## # A tibble: 3 × 3
## subject mass height
## <chr> <dbl> <dbl>
## 1 1 75 154
## 2 2 70 172
## 3 3 55 144现在数据tidy了,你可以使用ggplot(),问题得以解决。于是我们得出了一个结论:想要ggplot工作就需要tidy data。 如果这样想,那么今天的内容ggplot2统计图层就更加有必要了。
27.2 为何及何时使用统计图层
你可能每天都在用ggplot,却用不到stat_*()函数,这样也可以胜任很多工作。事实上,因为我们仅仅只使用geom_*()函数,你会发现stat_*()是开发者才使用的深奥和神秘的部分,如果这样想,你可能怀疑你是否有必要了解这些stat_*()函数。
好吧,学习 STAT 最主要的原因
“Even though the data is tidy, it may not represent the values you want to display”
我们这里再用一个例子说明,假定我们有一数据框simple_data
simple_data <- tibble(group = factor(rep(c("A", "B"), each = 15)),
subject = 1:30,
score = c(rnorm(15, 40, 20), rnorm(15, 60, 10)))
simple_data## # A tibble: 30 × 3
## group subject score
## <fct> <int> <dbl>
## 1 A 1 29.4
## 2 A 2 53.3
## 3 A 3 47.4
## 4 A 4 26.6
## 5 A 5 45.3
## 6 A 6 11.1
## 7 A 7 30.6
## 8 A 8 62.9
## 9 A 9 46.2
## 10 A 10 29.1
## # ℹ 20 more rows假定我们现在想画一个柱状图,一个柱子代表每一组group,柱子的高度代表的score的均值。
好比,按照我们的想法,我们首先规整(tidy)数据,并且确保数据包含每个geom所需的美学映射,最后传递给ggplot()
simple_data %>%
group_by(group) %>%
summarize(
mean_score = mean(score),
.groups = 'drop'
) %>%
ggplot(aes(x = group, y = mean_score)) +
geom_col()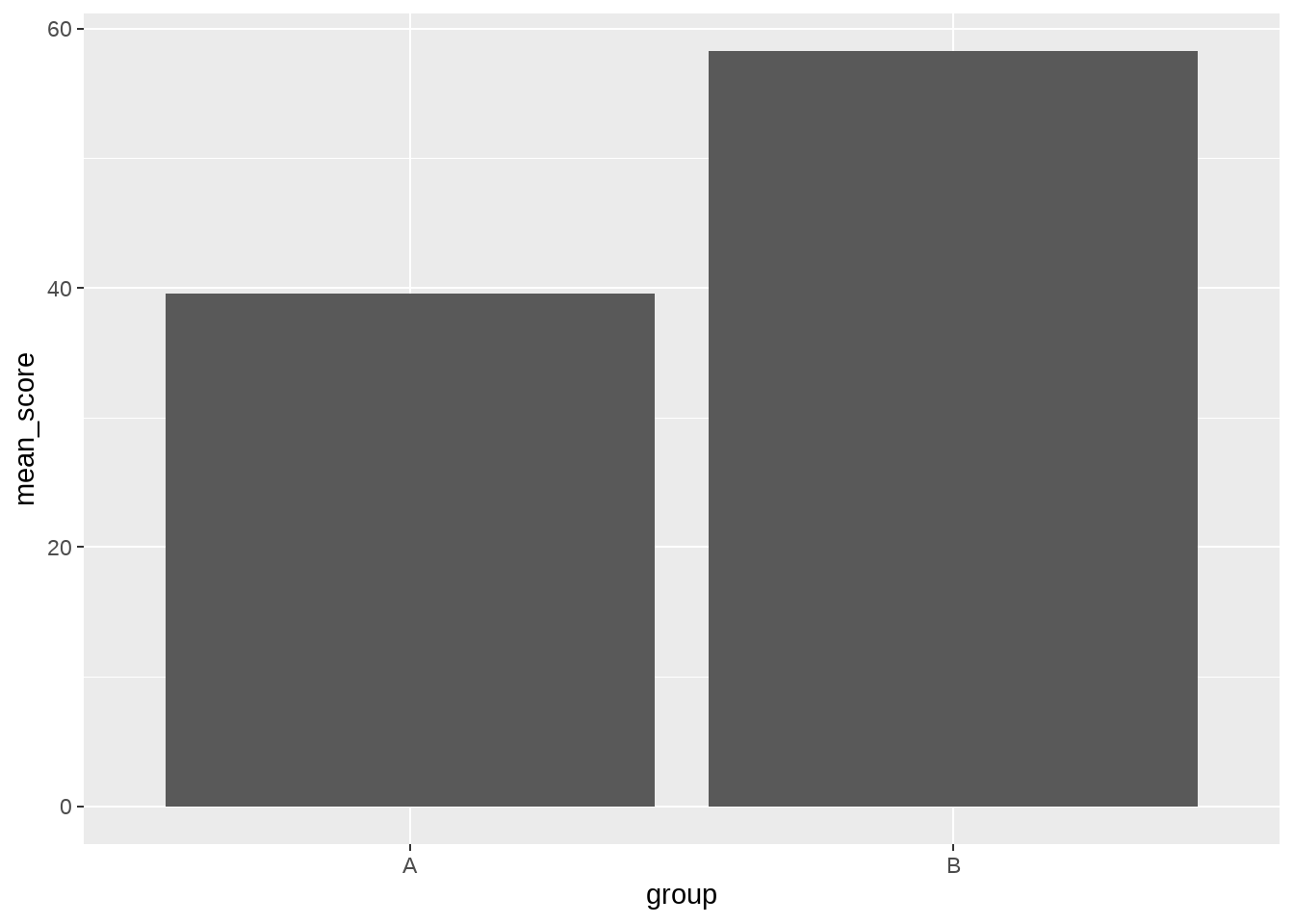
那么,传递给ggplot()的数据是
## # A tibble: 2 × 2
## group mean_score
## <fct> <dbl>
## 1 A 39.5
## 2 B 58.2需求很简单,很容易搞定。但如果我们想加误差棒(stand error)呢? 那我们需要再对数据整理统计,然后再传给ggplot().
于是,我们再计算误差棒,这里变型的数据是这个样子的
simple_data %>%
group_by(group) %>%
summarize(
mean_score = mean(score),
se = sqrt(var(score)/length(score)),
.groups = 'drop'
) %>%
mutate(
lower = mean_score - se,
upper = mean_score + se
)## # A tibble: 2 × 5
## group mean_score se lower upper
## <fct> <dbl> <dbl> <dbl> <dbl>
## 1 A 39.5 4.20 35.3 43.8
## 2 B 58.2 2.79 55.5 61.0然后把变型的数据传递给ggplot()
simple_data %>%
group_by(group) %>%
summarize(
mean_score = mean(score),
se = sqrt(var(score)/length(score)),
.groups = 'drop'
) %>%
mutate(
lower = mean_score - se,
upper = mean_score + se
) %>%
ggplot(aes(x = group, y = mean_score, ymin = lower, ymax = upper)) +
geom_errorbar()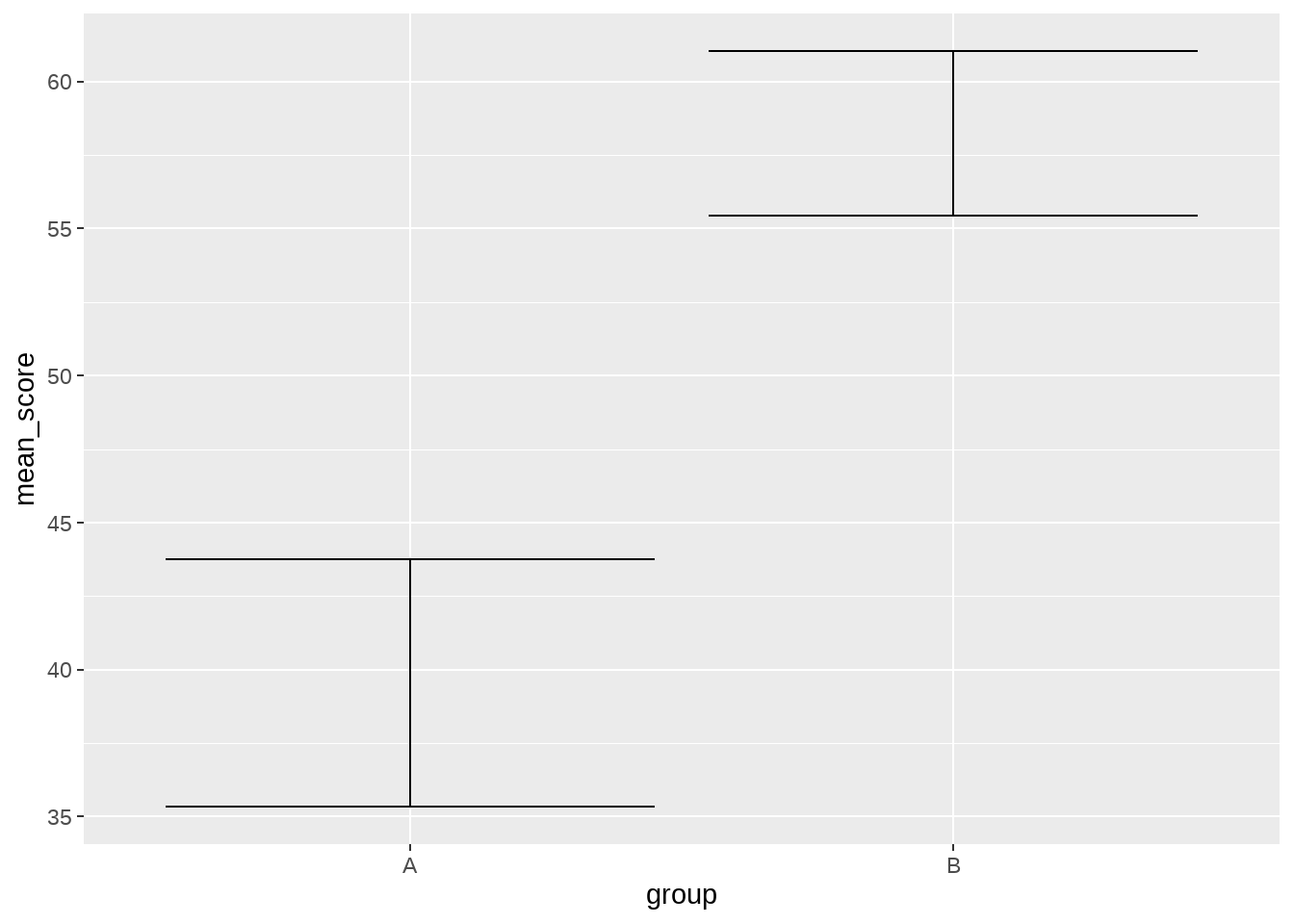
最后,我们把两个数据框组会到一起,一个用于柱状图,一个用于画误差棒。
simple_data_bar <- simple_data %>%
group_by(group) %>%
summarize(
mean_score = mean(score),
.groups = 'drop'
)
simple_data_errorbar <- simple_data %>%
group_by(group) %>%
summarize(
mean_score = mean(score),
se = sqrt(var(score)/length(score)),
.groups = 'drop'
) %>%
mutate(
lower = mean_score - se,
upper = mean_score + se
)
ggplot() +
geom_col(
aes(x = group, y = mean_score),
data = simple_data_bar
) +
geom_errorbar(
aes(x = group, y = mean_score, ymin = lower, ymax = upper),
data = simple_data_errorbar
)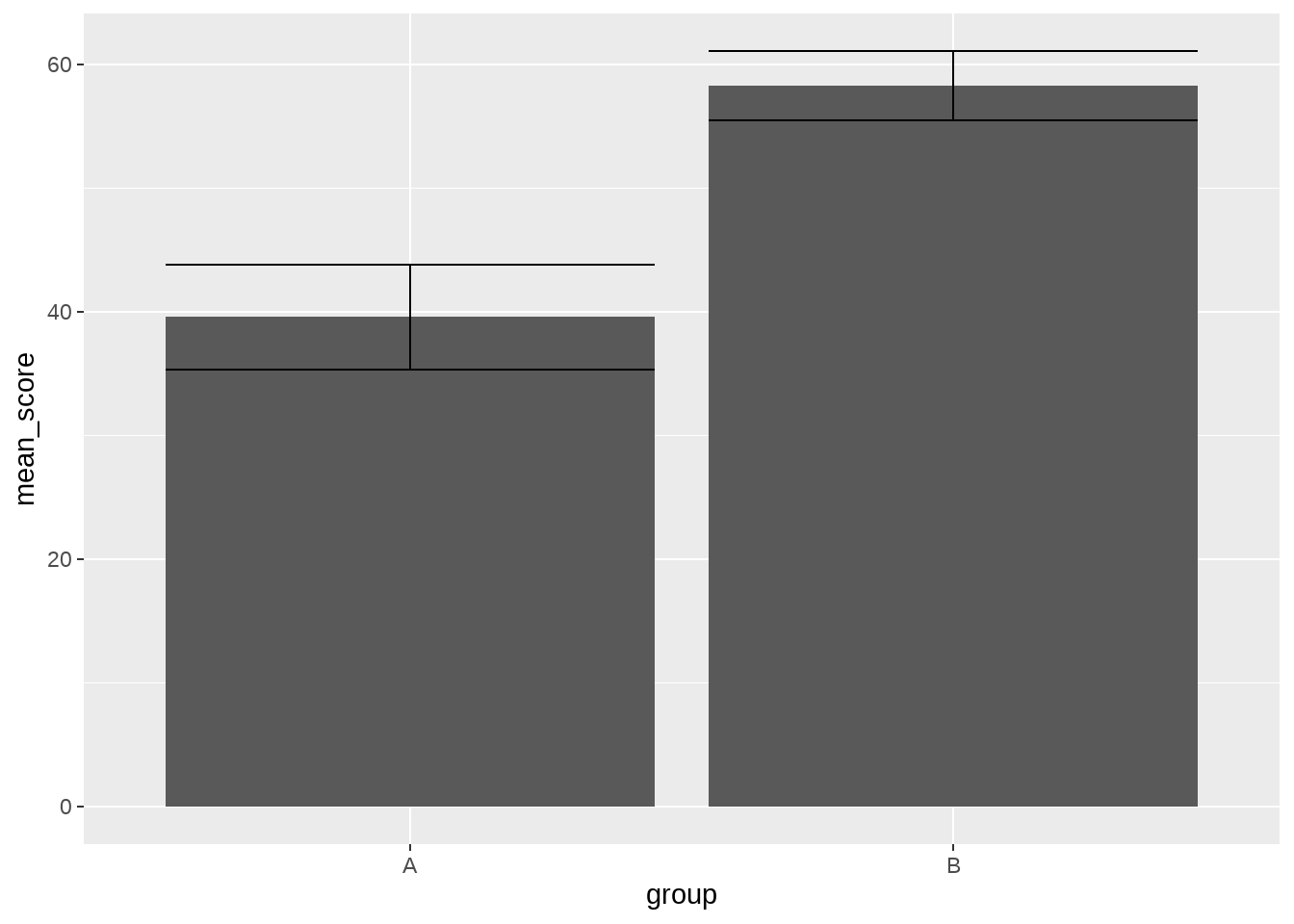
OMG, 为了画一个简单的图,我们需要写这么长的一段代码。究其原因就是,我们认为,一定要准备好一个tidy的数据,并且把想画的几何形状所需要的美学映射,都整理到这个tidy的数据框中
事实上,理论上讲,simple_data_bar 和 simple_data_errorbar 并不是真正的tidy格式。因为按照Hadley Wickham的对tidy的定义是,一行代表一次观察。
而这里的柱子的高度以及误差棒的两端不是观察出来的,而是统计计算出来的。
所以我们的观点是,辛辛苦苦创建一个(包含每个几何形状所需的美学映射)的数据框,太低效了,而且这种方法也不支持tidy原则。
既然 simple_data_bar 和 simple_data_errorbar都来源于simple_data,那为何不直接传递simple_data给ggplot(),让数据在内部转换,得到每个几何形状所需的美学映射呢?
或许,你想要的是这样?
simple_data %>%
ggplot(aes(group, score)) +
stat_summary(geom = "bar") +
stat_summary(geom = "errorbar")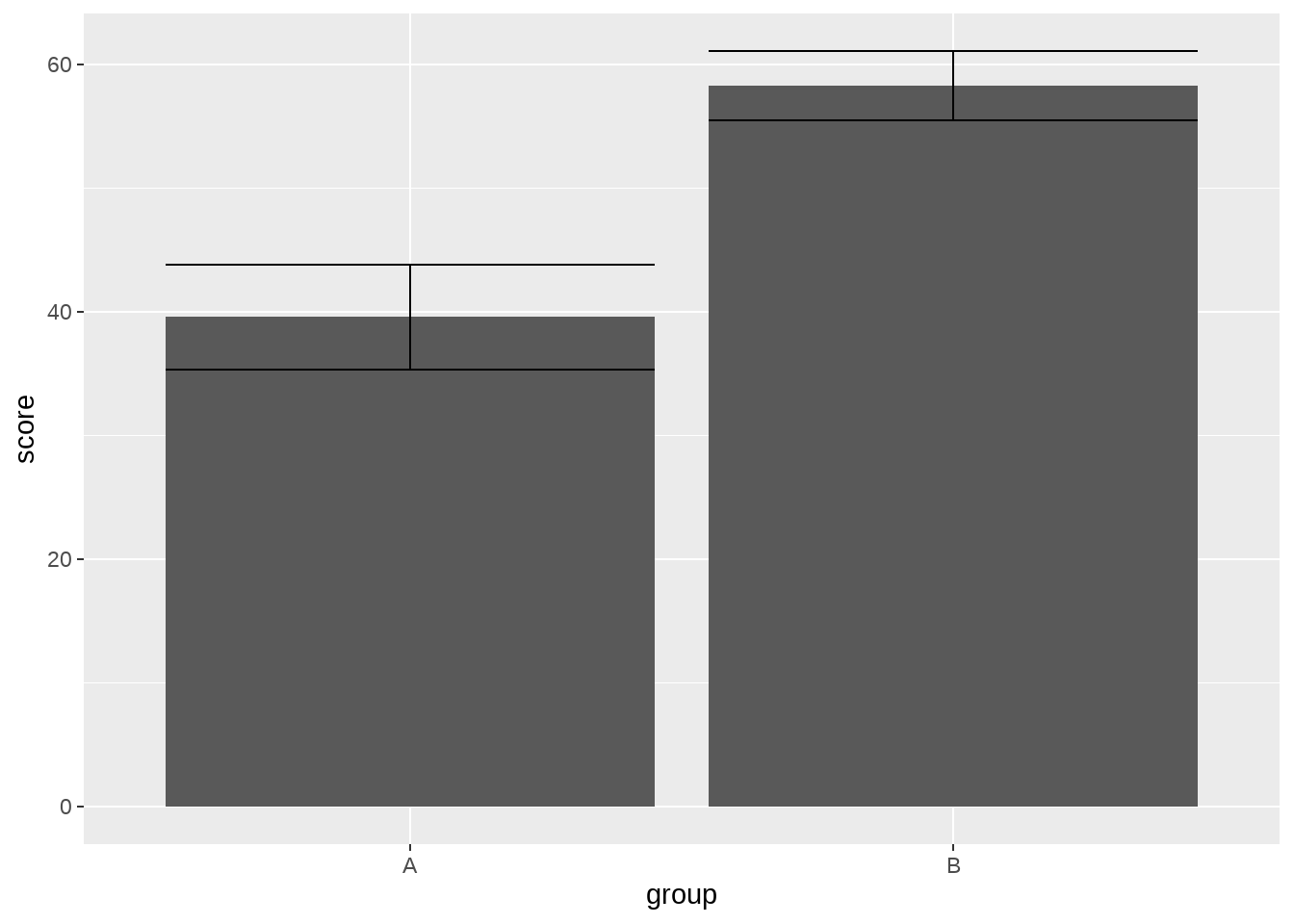
Bingo
27.2.1 小结
这一节,我们用一个很长的数据整理的代码,借助geom_*()画了一张含有误差棒的柱状图,而用stat_summary()不需要数据整理,只需要两行代码就实现相同效果。
感受到了stat_summary()的强大了?
不忙,好戏才慢慢开始…
27.3 用 stat_summary() 理解统计图层
前面讲到的 stat_summary() 是学习和理解 stat_*() 很好的例子,理解了stat_summary()的工作原理,其它的stat_*()也就都明白了,
事实上,stat_summary()也是在数据视化中最常用的,因此我们接着讲它。
那么,我们现在模拟一个测试数据height_df
用我们熟悉的geom_point()
height_df %>%
ggplot(aes(x = group, y = height)) +
geom_point()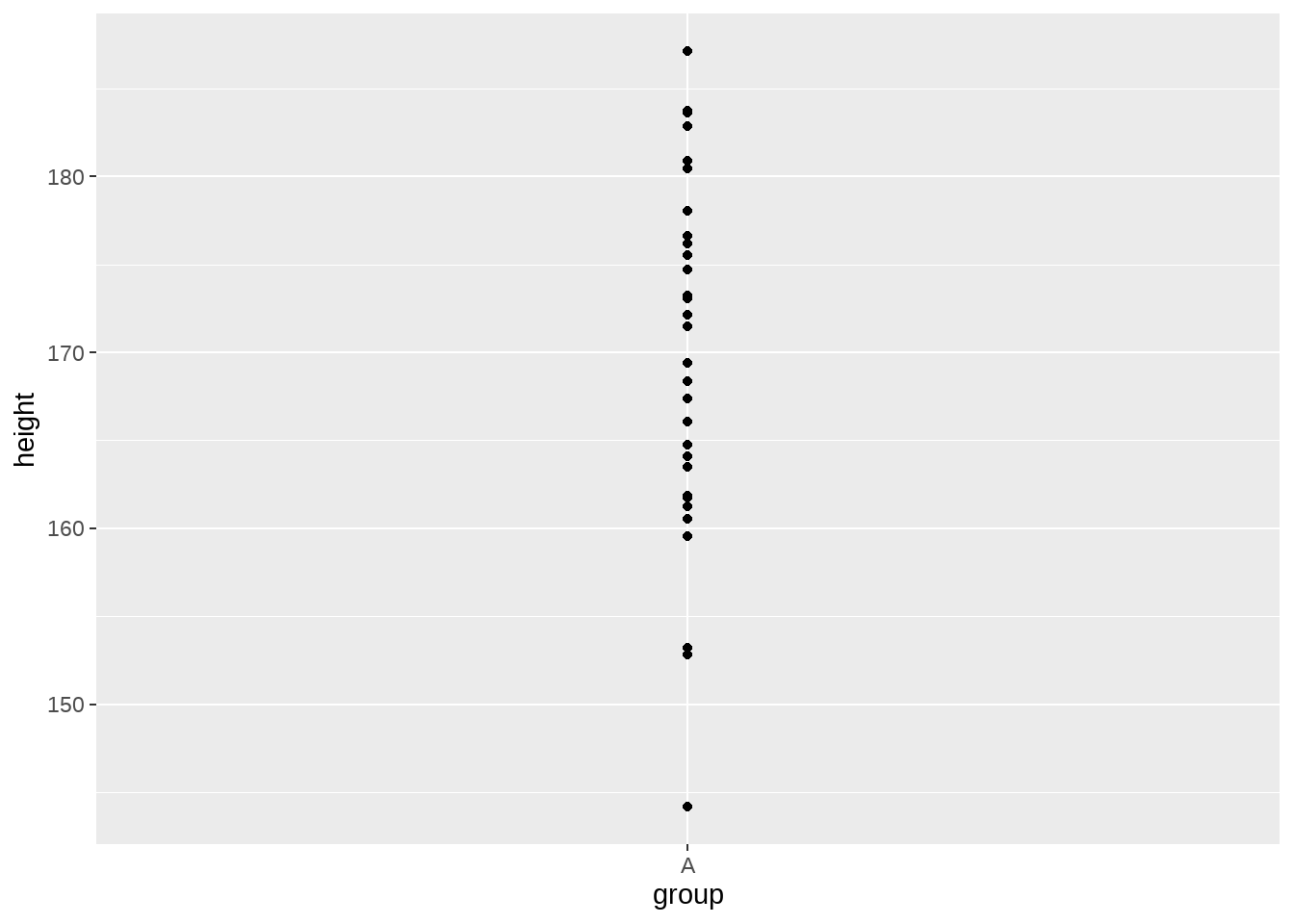
然后用stat_summary()代替geom_point(),然后看看发生了什么
height_df %>%
ggplot(aes(x = group, y = height)) +
stat_summary()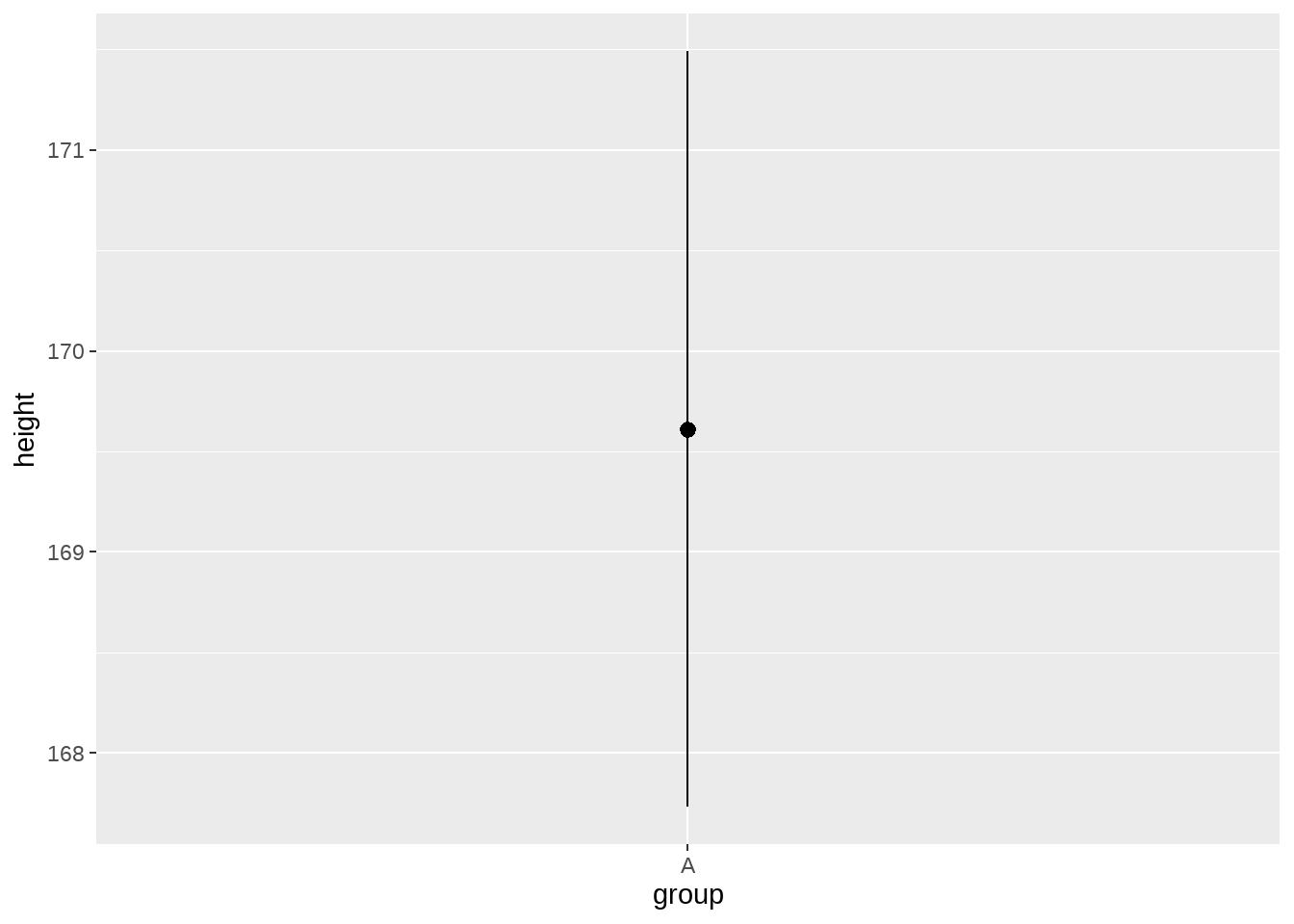
看到了一个点和经过这个点的一条线,实际上,它也是一个几何形状pointrange.
那么geom_pointrange() 是怎么数据转换的呢?回答这个问题,我们需要了解下geom_pointrange()需要哪些美学映射(参见图 27.1):
- x or y
- ymin or xmin
- ymax or xmax
所以,我们回去看看ggplot(aes(x = group, y = height))中aes()里的参数,group 映射到 x, height映射到了y, 但我们没有发现有ymin / xmin或者ymax / xmax的踪迹。问题来了,我们没有给出geom_pointrange()需要的美学映射,那stat_summmary()是怎么画出pointrange的呢?
我们先猜测一下,stat_summary()先计算出必要的数据值,然后传递给pointrange?
是不是呢?我们先看上图过程中有个提示
看到了吧,summary function,说明我们猜对了,这就是stat_*()神秘的地方。
- 首先,对于
stat_summary()中的fun.data参数,它的默认值是mean_se() - 其次,我们看看这个函数
mean_sefunction (x, mult = 1)
{
x <- stats::na.omit(x)
se <- mult * sqrt(stats::var(x)/length(x))
mean <- mean(x)
new_data_frame(list(y = mean, ymin = mean - se, ymax = mean +
se), n = 1)
}
<bytecode: 0x0000021aef28aa10>
<environment: namespace:ggplot2>这个mean_se()函数有两个参数,一个是x,一个是mult(默认为1), 那么这个函数的功能,一步步来说
- 删除缺失值
NA - 计算出
se, 公式为SE=√1N∑Ni=1(xi−ˉx)2 - 计算
x的均值 - 创建一个数据框(一行三列),
y = mean, ymin = mean - se, ymax = mean + se
很酷的一件事情是,mean_se()看上去是在ggplot()内部使用,实际上加载ggplot2宏包后,在全局环境变量里就可以访问到,不妨试试看, 注意到stat_summary()是对向量(单维度)做统计,因此要传height_df$height给它
mean_se(height_df$height)## y ymin ymax
## 1 169.6118 167.7341 171.4894数据看上去和我们前面 stat_summary() 画的点线图一样。当然为了保险起见,我们还是核对下,这里用到ggplot2包中的一个神奇的函数layer_data(), 它可以拉取在图层中使用的数据,第二个参数是指定拉取哪个图层的数据,这里只有唯一的一个图层,因此指定为1。
pointrange_plot <- height_df %>%
ggplot(aes(x = group, y = height)) +
stat_summary()
layer_data(pointrange_plot, 1)## x group y ymin ymax PANEL flipped_aes colour size linewidth
## 1 1 1 169.6118 167.7341 171.4894 1 FALSE black 0.5 0.5
## linetype shape fill alpha stroke
## 1 1 19 NA NA 1喔喔,结果很丰富,我们注意到y, ymin, and ymax 的值与 mean_se() 计算的结果一致。
27.3.1 小结
我们揭开了stat_summary()统计图层的神秘面纱的一角:
- 函数
stat_summary()里若没有指定数据,那就会从ggplot(data = .)里继承 - 参数
fun.data会调用函数将数据变形,这个函数默认是mean_se() -
fun.data返回的是数据框,这个数据框将用于geom参数画图,这里缺省的geom是pointrange - 如果
fun.data返回的数据框包含了所需要的美学映射,图形就会显示出来。
为了让大家看的更明白,我们在stat_summary()中显式地给出fun.data和geom两个参数
height_df %>%
ggplot(aes(x = group, y = height)) +
stat_summary(
geom = "pointrange",
fun.data = mean_se
)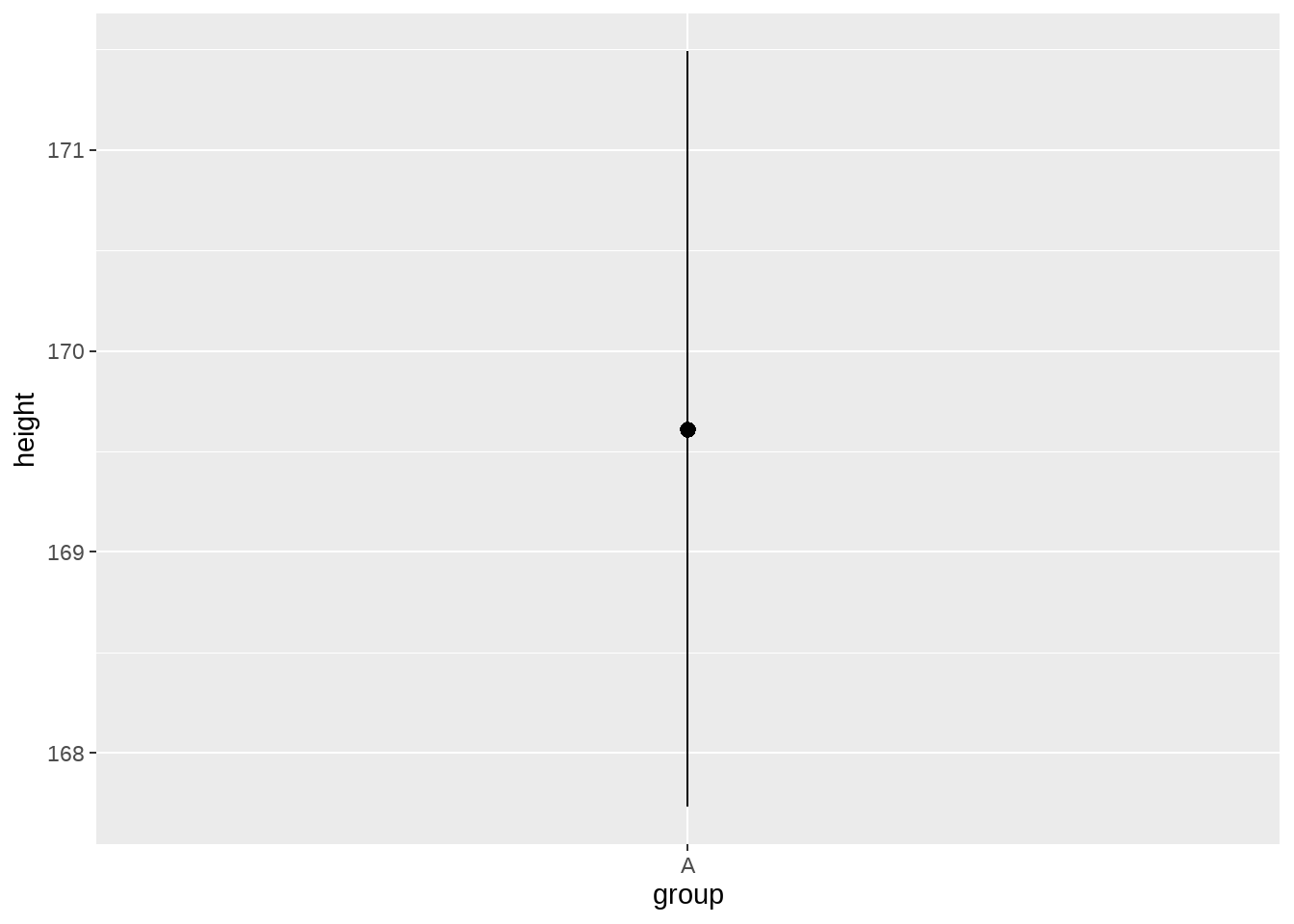
Look, it’s the same plot!
27.4 使用统计图层
现在我们进入了stat_summary()有趣的环节: 调整其中的参数画出各种图
27.4.1 包含95%置信区间的误差棒
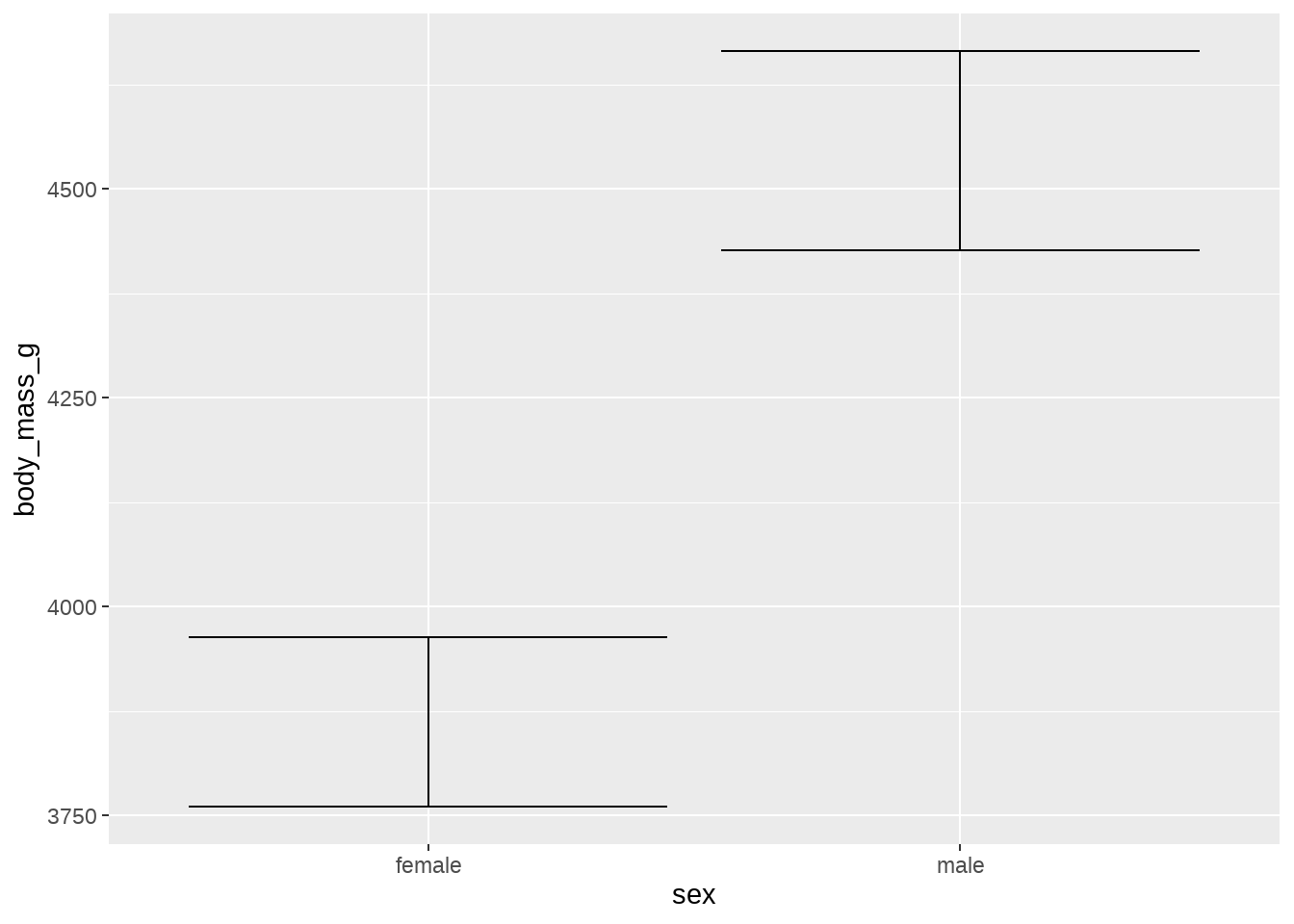
我们用企鹅数据画出不同性别sex下的企鹅体重均值,同时误差棒要给出95%的置信区间( 即均值加减 1.96倍的标准误)
my_penguins <- na.omit(penguins)
my_penguins %>%
ggplot(aes(sex, body_mass_g)) +
stat_summary(
fun.data = ~mean_se(., mult = 1.96), # Increase `mult` value for bigger interval!
geom = "errorbar",
)
那么这里在stat_summary()函数内部发生了什么呢?
分组分别各自的mean_se(),
female_mean_se <- my_penguins %>%
filter(sex == "female") %>%
pull(body_mass_g) %>%
mean_se(., mult = 1.96)
male_mean_se <- my_penguins %>%
filter(sex == "male") %>%
pull(body_mass_g) %>%
mean_se(., mult = 1.96)
bind_rows(female_mean_se, male_mean_se)## y ymin ymax
## 1 3862.273 3760.624 3963.921
## 2 4545.685 4426.581 4664.788当ggplot()中提供了分组变量(比如这里的sex),stat_summary()会分组计算,
再次感受到ggplot2的强大气息!
27.4.2 带有彩色填充色的柱状图
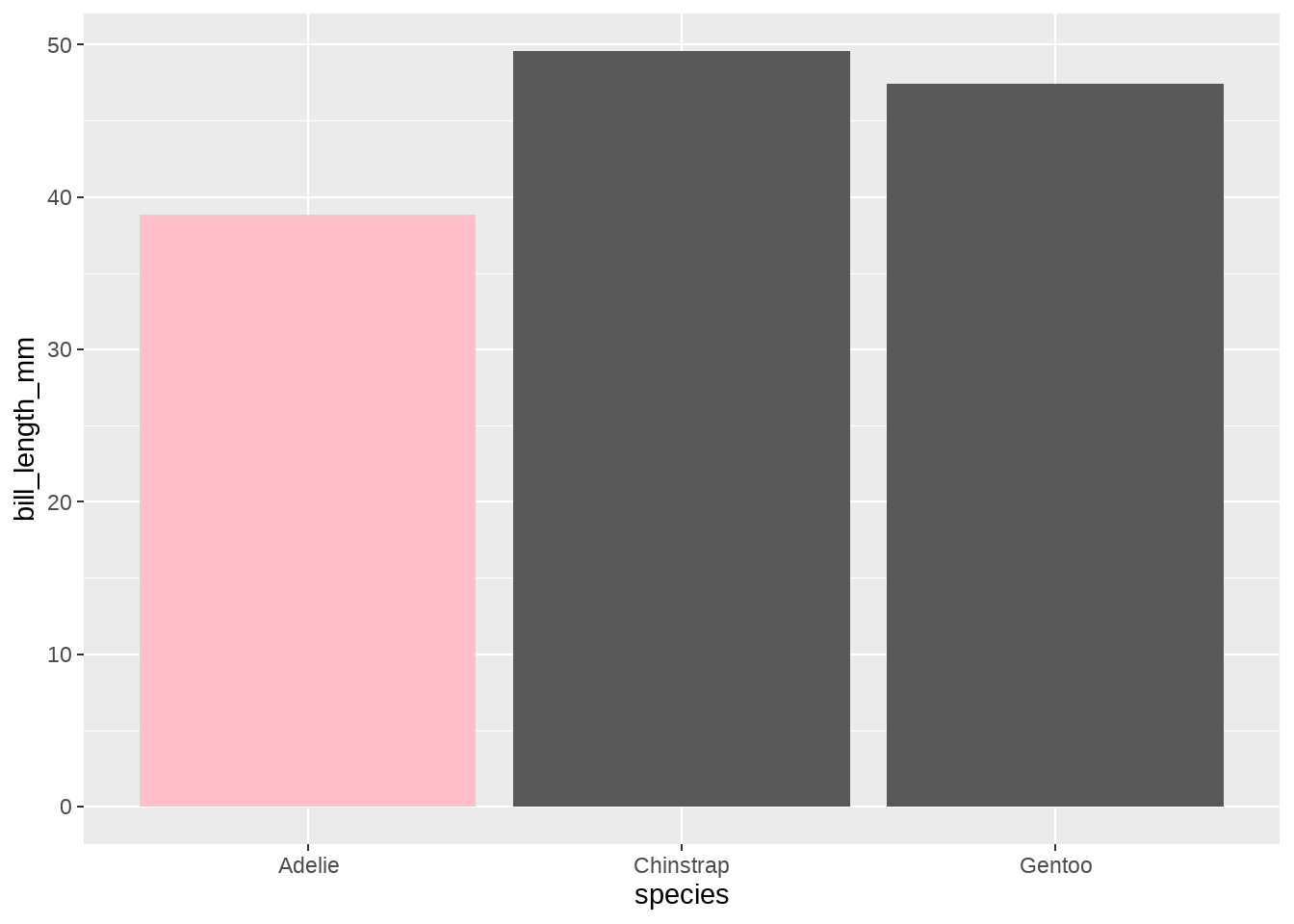
不同的企鹅种类,画出bill_length_mm长度的中位数(不再是均值),同时,让中位数小于40的用粉红色标出。这里需要自定义fun.data函数
calc_median_and_color <- function(x, threshold = 40) {
tibble(y = median(x)) %>%
mutate(fill = ifelse(y < threshold, "pink", "grey35"))
}
my_penguins %>%
ggplot(aes(species, bill_length_mm)) +
stat_summary(
fun.data = calc_median_and_color,
geom = "bar"
)
我们再来看看,stat_summary()内部发生了什么?
my_penguins %>%
group_split(species) %>%
map(~ pull(., bill_length_mm)) %>%
map_dfr(calc_median_and_color)## # A tibble: 3 × 2
## y fill
## <dbl> <chr>
## 1 38.8 pink
## 2 49.6 grey35
## 3 47.4 grey35注意到,fun.data中的定制函数还可以计算fill美学映射,最后一起传递给geom画图,强大!
27.4.3 大小变化的点线图
我们现在想画不同岛屿islands上企鹅bill_depth_mm均值,要求点线图中点的大小随观测数量(该岛屿企鹅的数量)变化
my_penguins %>%
ggplot(aes(species, bill_depth_mm)) +
stat_summary(
fun.data = function(x) {
scaled_size <- length(x)/nrow(my_penguins)
mean_se(x) %>%
mutate(size = scaled_size)
}
)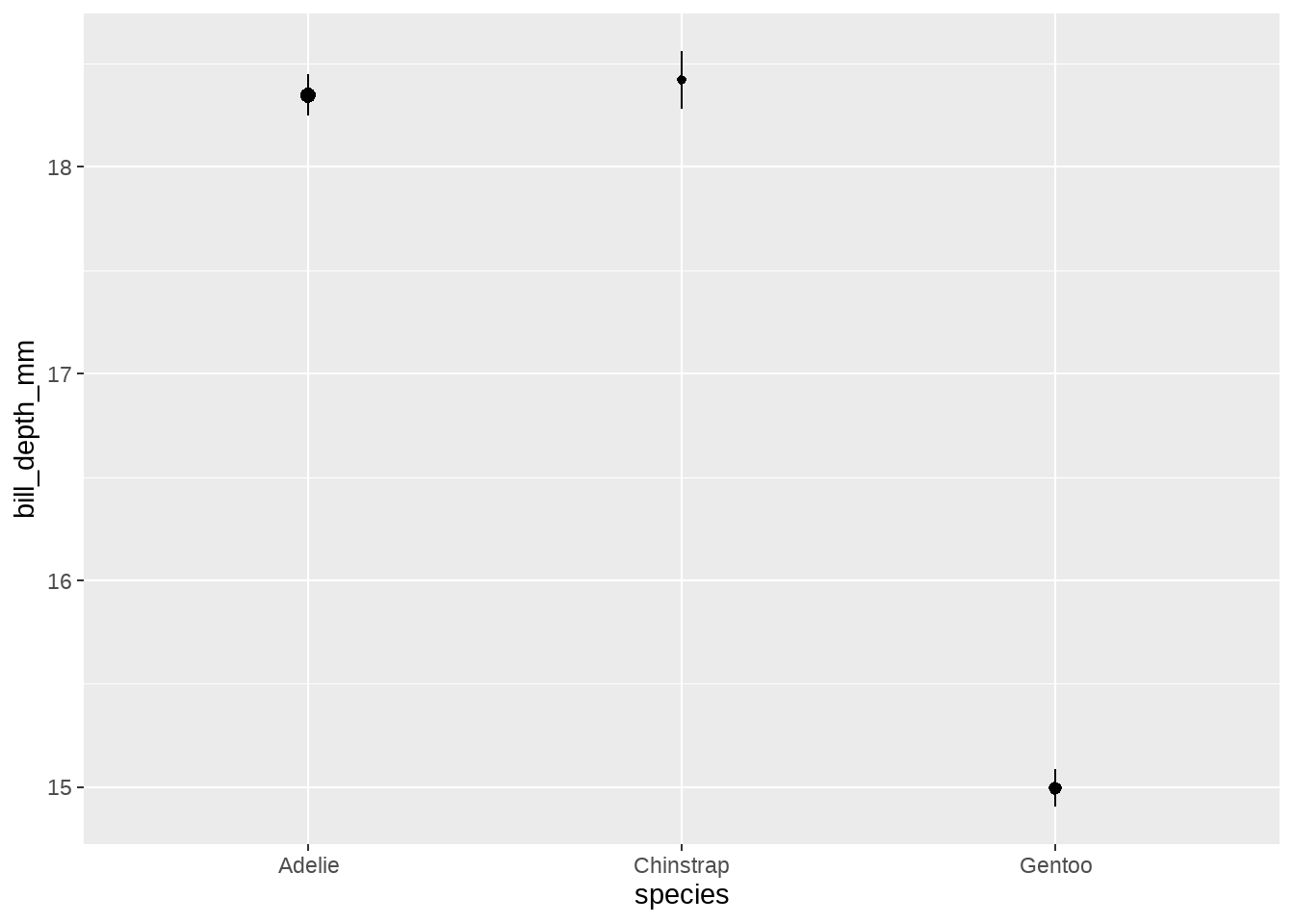
这张图其实听酷的,每个岛屿观察值越小(也就说样本量越小),pointrange的不确定性就越大(图中的误差棒范围就越长)。我们再看看,这里的stat_summary()内部发生了什么,或者说数据是怎么转换的。
my_penguins %>%
group_split(species) %>%
map(~ pull(., bill_depth_mm)) %>%
map_dfr(
function(x) {
scaled_size <- length(x)/nrow(my_penguins)
mean_se(x) %>%
mutate(size = scaled_size)
}
)## y ymin ymax size
## 1 18.34726 18.24635 18.44817 0.4384384
## 2 18.42059 18.28290 18.55828 0.2042042
## 3 14.99664 14.90625 15.08702 0.357357427.5 总结
27.5.1 主要结论
尽管数据是tidy的,但它未必能代表你想展示的值
解决办法不是去规整数据以符合几何形状的要求,而是将原初tidy数据传递给
ggplot(), 让stat_*()函数在内部实现变型可以
stat_*()函数可以定制geom以及相应的变形函数。当然,定制自己的函数,需要核对stat_*()所需要的变量和数据类型如果想用不同的geom,确保变换函数能计算出(几何形状所需要的)美学映射
27.5.2 STAT vs. GEOM or STAT and GEOM?
尽管我们在谈论geom_*()的局限性,从而衬托出stat_*()的强大,但并不意味了后者可以取代前者,因为这不是一个非此即彼的问题,事实上,他们彼此依赖– 我们看到stat_summary() 有 geom 参数, geom_*() 也有 stat 参数。
在更高的层级上讲,stat_*()和 geom_*() 都只是ggplot里构建图层的layer()函数的一个便利的方法,用曹植的《七步诗》来说, 本是同根生,相煎何太急。
将layer()分成stat_*()和 geom_*()两块,或许是一个失误,最后我们用Hadley的原话来结束本章内容
Unfortunately, due to an early design mistake I called these either stat_() or geom_(). A better decision would have been to call them layer_() functions: that’s a more accurate description because every layer involves a stat and a geom